巧合太多了,就不再是巧合了。
接续:新冠中的跨物种、跨生物界、超生物基因 3
I SARS-CoV-2对冠状病毒基因的继承、借鉴、模仿、优化、集成(续三)
MHV-JHM、MHV-3、FIPV、FECV,及S2/S2处的多重借鉴(续)
3、S1/S2处的多重借鉴
SARS-CoV-2 S1/S2位点附近的氨基酸序列和核苷酸序列是多方借鉴、精心设计的产物。多重借鉴设计使SARS-CoV-2兼具不同借鉴来源的多重感染、致病、生存和人体适应优势。
S1/S2表示冠状病毒Spike蛋白S1亚基、S2亚基交界处。
第一重借鉴:RRARR借鉴
如上一篇文章所述,SARS-CoV-2 S1/S2处的基本furin切割序列RRAR借鉴了小鼠冠状病毒MHV-JHM株系、MHV-3株系,及猫冠状病毒(FIPV、FECV)某些病毒株等病毒S1/S2处的furin切割序列RRARR(、RRAR)。
第二重借鉴:人类ENaC-α长furin切割序列借鉴
SARS-CoV-2 S1/S2处的长furin切割序列RRAR'SVAS借鉴、复用了人类ENaC-α的长furin切割序列。
SARS-CoV-2 S1/S2处基本furin切割序列RRAR的四个氨基酸,与右侧紧邻的四个氨基酸SVAS,共八个氨基酸组成的序列RRARSVAS(精氨酸-精氨酸-丙氨酸-精氨酸-丝氨酸-缬氨酸-丙氨酸-丝氨酸)称为 SARS-CoV-2的长furin切割序列。为方便研究每一个位点,通常将长furin切割序列的八个氨基酸位点从左到右依次标记为P4、P3、P2、P1、P1'、P2'、P3'、P4'。上一篇文章展示猫冠状病毒FIPV 、FECV S1/S2处furin切割序列的两个配图示范了这种标记方式。
RRARSVAS也可表示为RRAR'SVAS,中间所加的分隔符“'”既代表S1、S2亚基的分界线,也指示着furin蛋白酶的切割位置。
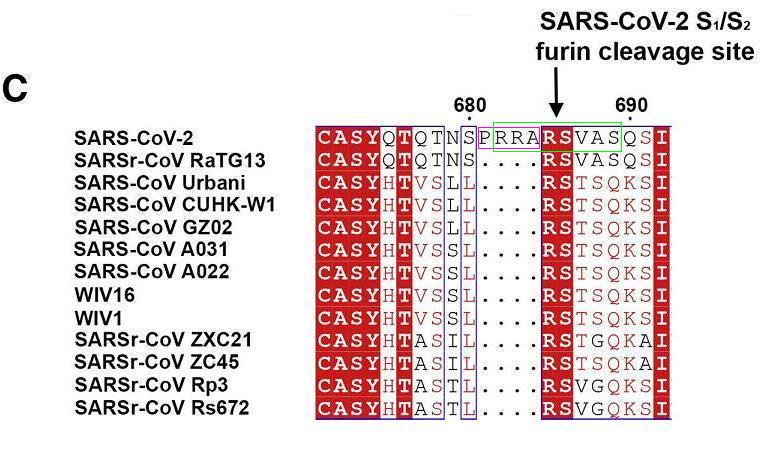
SARS-CoV-2 S1/S2处特有的PRRA插入、基本fuin切割序列RRAR、长fuin切割序列RRARSVAS
SARS-CoV-2 S1/S2处的长furin切割序列,与人类ENaC-α的长furin切割序列,两个aa序列(amino acid sequence,氨基酸序列)完全相同,皆为RRAR'SVAS,如下图b所示:
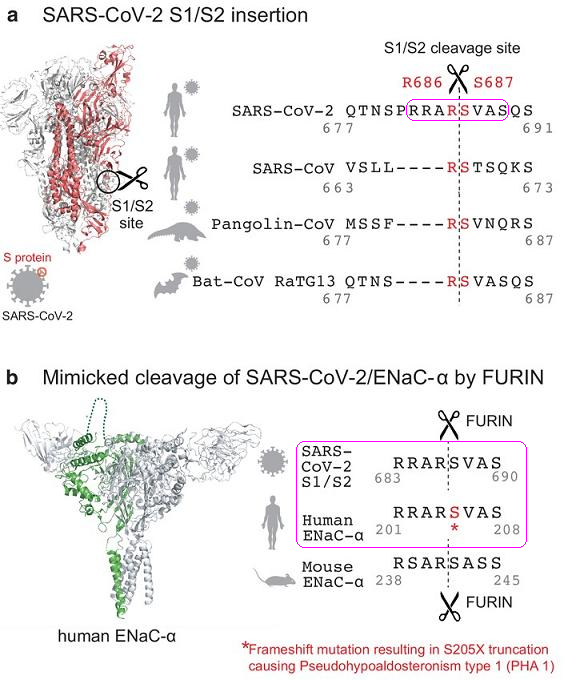
图a:SARS-2、三种SARS-2 “近亲”S1/S2位点附近的aa序列对照;
图b:SARS-2 Spike、人类ENaC-α、小鼠ENaC-α的长furin切割序列对照。
论文依据:
SARS-CoV-2 strategically mimics proteolytic activation of human ENaC
https://elifesciences.org/articles/58603
A call for an independent inquiry into the origin of the SARS-CoV-2 virus
https://www.pnas.org/doi/10.1073/pnas.2202769119
ENaC-α是ENaC的α亚基;ENaC是Epithelial Na+ Channel(上皮钠离子通道)的缩写,也叫做Epithelial Sodium Channel (上皮钠通道),是控制钠离子进出细胞膜的跨膜离子通道。ENaC存在于人类和脊椎动物的肺(肺泡)、呼吸道(鼻腔、气管、支气管)、口腔(舌角质形成细胞)、肾脏、结肠、胰腺、汗腺、唾液腺等众多器官、结构的上皮组织中,对维持器官、组织的盐水平衡、水稳态和正常生理功能至关重要。一些ENaC高丰度分布的部位,如鼻腔上皮、上下呼吸道上皮、肺泡上皮,恰恰是SARS-CoV-2的首要感染部位;此外,ENaC、ACE2受体两者在人体内的分布部位高度重合。
SARS-CoV-2为什么要借鉴、复用人类ENaC-α的长furin切割序列RRAR'SVAS呢?
从某个技术视角看,P2位点不是碱性残基R或K(精氨酸或赖氨酸),而是非碱性残基A(丙氨酸)的RRAR不是最优的furin切割序列。有些人据此判断,SARS-CoV-2的基本furinfurin切割序列RRAR不是一个最优的设计,这表明SARS-CoV-2不是人为设计的。如果SARS-CoV-2出于人为设计,那么,设计者不应使用一个不够好的furin切割设计方案。SARS-CoV-2的切割序列真的不够好吗?SARS-CoV-2自然来源论的维护者们是离不开选择性遮蔽式举证的,他们不敢让你知道全部事实,这次他们又忽略或有意回避了一个关键事实:RRAR'SVAS是人类ENaC-α的furin切割序列。这一事实决定了,RRAR'SVAS恐怕是用于对人类感染、致病的最佳furin切割序列,这体现在:
1)、RRAR'SVAS能兼容包括furin在内的众多人体蛋白酶,拥有RRAR'SVAS序列的SARS-CoV-2 Spike蛋白能够被包括furin在内的众多人体蛋白酶高效水解、激活。
2)、RRAR'SVAS能充分利用ENaC、ACE2在人体内的分布情况,使SARS-CoV-2在上下呼吸道上皮、肺泡上皮等组织中能够顺利地同时完成受体结合和直接膜融合,从而实现高效细胞进入(即感染)。
3)、SARS-CoV-2对人类ENaC-α长furin切割序列的借鉴、复用使SARS-CoV-2的Spike蛋白能够天然适应人体的蛋白水解环境,得以模仿、伪装并最大程度利用人体ENaC-α的水解、激活机制,得以在人体各处组织中被包括furin蛋白酶在内的多种人体蛋白酶视作人体结构高效裂解、激活,高效感染人体细胞,在人体组织内、组织间高效扩散,并迷惑免疫系统,逃避免疫攻击。
注:SARS-CoV-2的FCS(furin cleavage site,弗林切割位点)可诱导人体细胞间发生膜融合,形成多细胞核合胞体-巨细胞,SARS-CoV-2可通过合胞体在组织内和人体相邻组织间扩散;SARS-CoV-2 Spike还可通过借鉴自昆虫多角体病毒(属于杆状病毒科)的几丁质结构域(Chitin-binding type-2)吸附红细胞,搭乘红细胞“便车”通过血液循环在人体内更高效地扩散,迷惑并逃避免疫攻击。
与人体ENaC竞争furin蛋白酶,或劫持ENaC所需的furin蛋白酶可能造成ENaC功能紊乱、失常。ENaC功能紊乱可能引发高血压,低血钾,肾脏水、钠潴留,代谢性碱中毒等病症,这些病症还可能与SARS-CoV-2的其它致病能力相互作用,引发更复杂、更严重的病症。
SARS-CoV-2感染,SARS-CoV-2与人体ENaC-α竞争furin蛋白酶可能导致furin蛋白酶异常激活及furin蛋白酶表达水平发生改变。furin蛋白酶异常激活和表达水平的改变可能导致癌症和神经退行性疾病(neurodegenerative disorder)等疾病。笔者已多次指出,SARS-CoV-2 具有高度致癌性,SARS-CoV-2的致癌途径非常丰富。
RRAR'SVAS序列在生物界、微生物界、病毒界都极为罕见,这体现在:
1)、SARS-CoV-2是唯一一种Spike蛋白含有RRAR'SVAS序列的冠状病毒,SARS-CoV-2外的所有冠状病毒Spike蛋白皆不含RRAR'SVAS序列。
2)、ENaC-α是aa序列(氨基酸序列)含有RRAR'SVAS的唯一一种人体结构,ENaC-α之外的其它人体结构皆不含有RRAR'SVAS序列;
3)、只有六个物种的ENaC-α含有RRAR'SVAS序列,其中五种是灵长类动物:人类、黑猩猩、矮黑猩猩、猩猩、大猩猩,第六种,也是唯一一种ENaC-α含有RRAR'SVAS序列的非人类、非灵长类物种是存在于欧洲和西亚的一种叫作pipistrellus kuhlii的蝙蝠。
第1)、2)两点是以下论文的生物数据库搜索匹配结论:
SARS-CoV-2 strategically mimics proteolytic activation of human ENaC
(SARS-CoV-2策略性地模拟人类ENaC的蛋白水解激活)
https://elifesciences.org/articles/58603
论文原文:
Now,
Anand et al. show that the spike proteins on SARS-CoV-2 may have the
same sequence of amino acids at its cut site as a human epithelial
channel protein called EnaC-α.
…
SARS-CoV-2 contains an 8-mer
peptide that is identical to the furin cleavage site in the α subunit of
ENaC located at the C-terminal end of the putative inhibitory segment.
…
Hence,
the authors should better stress the novelty of their paper, which
resides in the correlation be-tween this cleavage site that is present
only in SARS-CoV-2 (and not in other coronaviruses) and the ENAC-α,
…
The
resulting tribasic 8-mer peptide (RRARSVAS) on the SARS-CoV-2 S1/S2
site is conserved among 10,956 of 10,967 circulating strains deposited
at GISAID (https://www.gisaid.org/) (Elbe and Buckland-Merrett, 2017),
as of April 28, 2020 (Supplementary file 1a). This peptide is also
absent in over 13,000 non-COVID-19 coronavirus S-proteins from the VIPR
database (Carrillo-Tripp et al., 2009). Strikingly, examining over 10
million peptides (8-mers) of 20,350 canonical human proteins from
UniProtKB shows that the peptide of interest (RRARSVAS) is present
exclusively in human ENaC-ɑ, also known as SCNN1A (p-value=4E-4) (see
Materials and methods).
第3)点是以下论文的生物库数据搜索、匹配结论:
A call for an independent inquiry into the origin of the SARS-CoV-2 virus
https://www.pnas.org/doi/10.1073/pnas.2202769119
论文原文:
The
FCS sequence of human ENaC α (20) is identical in chimpanzee, bonobo,
orangutan, and gorilla (SI Appendix, Fig. 1), but diverges in all other
species, even primates, except one. (The one non-human non-great ape
species with the same sequence is Pipistrellus kuhlii, a bat species
found in Europe and Western Asia; other bat species, including
Rhinolophus ferrumequinem, have a different FCS sequence in ENaC α
[RKAR'SAAS]).
可以说,SARS-CoV-2 Spike蛋白与人类ENaC-α具有相同的长furin切割序列RRAR'SVAS这一结构巧合,是匪夷所思的0概率事件。
此外,RRAR'SVAS序列有可能在其它冠状病毒或非冠状病毒的各蛋白中都不存在,很多论文做过生物数据库搜索、匹配研究,但未见有论文报告、指出,在某种非SARS-CoV-2病毒的某种蛋白中也存在RRAR'SVAS序列。
甚至,RRAR'SVAS序列有可能只存在于SARS-CoV-2的Spike蛋白和六个物种的ENaC-α之中,而不存在于其它任何生物结构、生物蛋白、病毒蛋白中。未见有论文报告,除ENaC-α外,何种其它生物结构、生物蛋白也含有RRAR'SVAS序列。
为什么偏偏是SARS-CoV-2“自然变异”、“自然演化”出了生物界极度罕见,病毒界独一无二,“恰好”与人类ENaC-α相同的长furin切割序列RRAR'SVAS?这一序列为什么极为精准地恰好出现在对感染、致病能力至关重要的S1/S2位点(SARS-CoV-2共有近3万个核苷酸位点)?SARS-CoV-2 S1/S2处的长furin切割序列RRAR'SVAS是随机性、无目的自然变异、自然重组、自然演化的产物,还是人为借鉴、人为设计的产物?
相关文章:
谁设计、制造了新冠病毒(四)拉尔夫·巴里克与肺研究所 1
谁设计、制造了新冠病毒(四)北卡肺研究所与ENaC
“SARS-CoV-2 Spike蛋白与人类ENaC-α具有相同的长furin切割序列”这一重大事实、重大巧合、重大铁证,自疫情发生至今,始终没有任何一家重量级媒体报道、告知公众。何为公众知情权?何为信息自由、信息透明?他们的势力庞大到了何种程度!他们心虚到了何种程度!
第三重借鉴:莫德纳专利序列借鉴
SARS-CoV-2 Spike蛋白与人类ENaC-α的长furin切割序列皆为RRAR'SVAS;不过,同一氨基酸序列RRAR'SVAS在SARS-CoV-2和人类ENaC-α中对应的核苷酸序列并不相同(同一氨基酸可以用不同的核苷酸密码子表达),RAR'SVAS序列所含8个氨基酸中的7个,在SARS-CoV-2和人类ENaC-α使用了不同的核苷酸密码子。表达氨基酸的3个核苷酸的组合称为氨基酸的密码子,一个氨基酸可能有多个密码子。
SARS-CoV-2 S1/S2位点附近的核苷酸序列另有借鉴:SARS-CoV-2 S1/S2位点附近与7氨基酸序列SPRRARS对应的19个核苷酸的序列借鉴自莫德纳专利中一段核苷酸序列的反向序列。
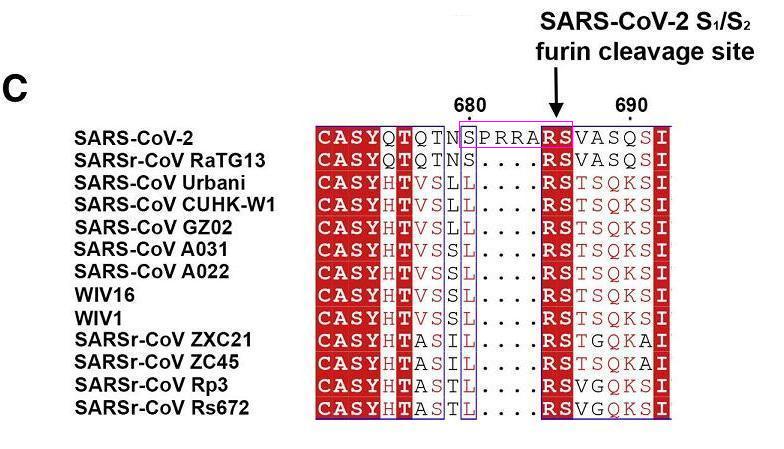
SARS-CoV-2 S1/S2位点附近与莫德纳专利对应的7氨基酸序列SPRRARS
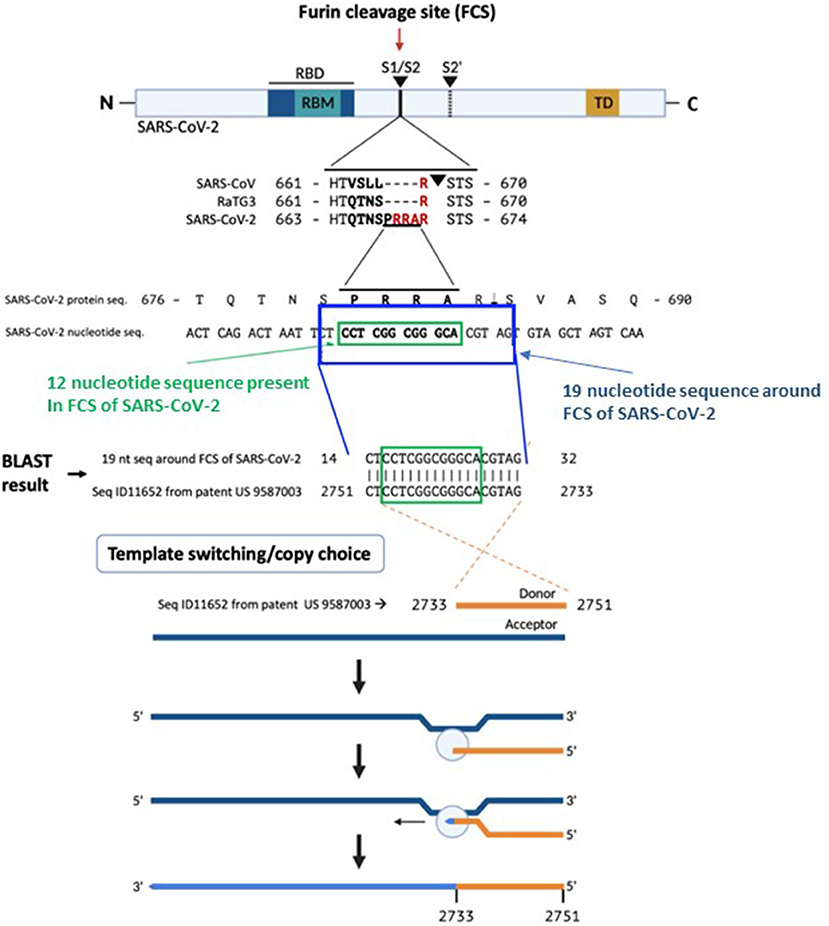
SARS-CoV-2 S1/S2处19核苷酸序列与莫德纳专利中核苷酸序列的匹配关系
如上图所示,SARS-CoV-2 S1/S2处的7氨基酸序列SPRRARS对应如下21核苷酸序列(一个氨基酸对应三个核苷酸):
TCT CCT CGG CGG GCA CGT AGT
A -腺嘌呤、G -鸟嘌呤、C-胞嘧啶、T-胸腺嘧啶。
去掉最左边和最右边的T后,得到如下19核苷酸序列:
CT CCT CGG CGG GCA CGT AG
莫德纳公司(Moderna Therapeutics, Inc)2016年申请的一项美国专利(patent US 9587003,Filed: February 4, 2016)中有一个长度约为3300的DNA核苷酸序列Seq ID11652,这个序列的第2733至2751共19个核苷酸倒过来写居然也是:
CT CCT CGG CGG GCA CGT AG
两个出处的19核苷酸序列完全相同!
以上是一个简明的描述,略去了若干细节,如RNA序列与DNA序列的互补匹配关系,且RNA核苷酸序列中的尿嘧啶U已被胸腺嘧啶T代替。下文中有更细致的说明:
新冠人造再添铁证,“有据核查”欲盖弥彰
指出以上“巧合”的是如下论文:
MSH3 Homology and Potential Recombination Link to SARS-CoV-2 Furin Cleavage Site
MSH3同源性及与SARS-CoV-2弗林切割位点的潜在重组联系
https://www.frontiersin.org/articles/10.3389/fviro.2022.834808/full
媒体报道过这篇论文。
莫德纳专利US 9587003是一项肿瘤相关的生物专利,专利名称及链接如下:
Modified polynucleotides for the production of oncology-related proteins and peptides
用于生产肿瘤相关蛋白质和多肽的修饰多核苷酸
https://uspto.report/patent/grant/9587003
https://patents.google.com/patent/US9587003B2/en
该专利中的SEQ ID11652是一个非天然序列,它与人类MSH3氨基酸序列100%相同,但核苷酸序列不同,是对人类MSH3序列加以密码子/核苷酸修饰/修改后得到的(在保持氨基酸无一改变的前提下,修改了部分氨基酸的核苷酸密码子)。如前所述,SARS-CoV-2 Spike蛋白的长furin切割序列与人类ENaC-α的长furin切割序列也是氨基酸序列完全相同,但核苷酸序列不同。
MSH3是人体蛋白mutS homolog 3的缩写,它是细菌DNA错配修复(mismatch repair,MMR)蛋白MutS的人类同源物,它的作用是纠正DNA合成过程中的碱基错配和缺失,许多肿瘤、癌症的发生与该蛋白基因突变或功能故障有关。在病毒中引入MSH3序列可能会干预、干扰人体的DNA错配修复(MMR)机制,引发人体细胞的错配修复缺陷,进而导致细胞癌变,并有利于病毒的感染、生存和致病能力。
莫德纳专利US 9587003中的SEQ ID11652序列可能引入了人和小鼠肿瘤中的核苷酸,或者说,SEQ ID11652可能修改了正常人类MSH3中氨基酸的核苷酸密码子,代之以肿瘤组织中MSH3氨基酸的核苷酸密码子。而来自肿瘤组织的核苷酸已被证明可使小鼠产生多种癌症。注意专利中的以下两段文字:
Lacour, F. et al., Transplantable malignant tumors in mice induced by preparations containing ribonucleic acid extracted from human and mouse tumors. J. Natl Cancer Inst., 1960, 24(2):301-27. cited by applicant .
Latarjet, R., Production of multiple cancers in mice having received nucleic acid extract from isologous & homologous leukemic tissues. C.R. Hebd Seances Acad. Sci., 1958, 246(5):853-5. cited by applicant .
这两段的机器译文:
Lacour, F. 等人,用含有从人和小鼠肿瘤中提取的核糖核酸的制剂诱发小鼠的可移植性恶性肿瘤。 J.国家癌症研究所,1960,24(2):301-27。 申请人引用。
Latarjet, R.,接受同源和同源白血病组织核酸提取物的小鼠产生多种癌症。 C.R. Hebd 降神会学院。 科学,1958,246(5):853-5。 申请人引用。(译文结束)
与MSH3的关系及序列来源都提示,莫德纳专利US 9587003中的SEQ ID11652序列可能是一个致癌序列;而SARS-CoV-2 S1/S2位点附近的19核苷酸序列借鉴、应用了该序列中一个19核苷酸子序列的反向序列。
SARS-CoV-2具有高度致癌性,SARS-CoV-2拥有非常丰富多样的致癌途径。
除SARS-CoV-2外,现有生物数据库记录的所有真核生物基因组或病毒基因组均不含有SARS-CoV-2与莫德纳专利共有的19核苷酸序列或其互补序列。该19核苷酸序列在某些细菌、古菌基因组中存在,细菌、古菌都不是真核生物。
前面指出的论文估算,莫德纳专利中的19核苷酸序列自然出现在SARS-CoV-2基因组中的概率是:3.21×10^-11。10的负11次方即千亿分之一,也就是说,按论文估算,莫德纳专利中的19核苷酸序列自然出现在SARS-CoV-2基因组中的概率约为千亿分之3.21。
论文的算法似乎并未考虑19核苷酸序列在SARS-CoV-2基因组中的出现位置,SARS-CoV-2共有约30000个核苷酸位点,因此,19核苷酸序列自然出现在SARS-CoV-2 S1/S2处的概率约为(3.21×10^-11)/30000≈(3.21×10^-11×10^-4)/3,即约1000万亿分之一。
第四重借鉴:MERS病毒S1/S2位点左侧氨基酸序列模式,及P5位点脯氨酸借鉴
SARS-CoV-2借鉴或沿用了MERS-CoV(中东呼吸综合症冠状病毒)S1/S2位点左侧氨基酸序列模式,以及P5位点的脯氨酸。
先回顾一下上一篇文章尾段的两个插图。
β谱系B支系部分人类、果子狸、貉、穿山甲、蝙蝠冠状病毒局部氨基酸序列比对
SARS-CoV-2(Wuhan-Hu-1毒株)与进化树中冠状病毒S1/S2附近的aa序列比对
由以上两图可见,SARS-CoV-2 S1/S2位点(S1、S2亚基交界处,第二个图红色箭头所示)左侧的四氨基酸PRRA在图中极为独特、突兀,与 SARS-CoV-2“演化”关系较近的冠状病毒无一例外,全都没有与PRRA 对应的氨基酸,这些病毒PRRA对应位点处的氨基酸,无一例外,全都空缺。
“演化”关系较近的病毒无一具有与PRRA对应的氨基酸,然而,与SARS-CoV-2 演化关系极远的MERS-CoV不仅具有与PRRA对应的氨基酸PRSV,而且二者非常相似。

四种病毒S1/S2位点附近的氨基酸序列

SARS-CoV-2、MERS-CoVS1/S2位点附近的氨基酸序列
如以上两图所示,SARS-CoV-2 S1/S2位点左侧四氨基酸为PRRA,MERS-CoV S1/S2位点左侧对应的四氨基酸为PRSV。PRRA促成了SARS-CoV-2的基本furin切割序列RRAR,PRSV则促成了MERS-CoV的基本furin切割序列RSVR 。PRRA、PRSV皆以PR(脯氨酸-精氨酸)开头,并皆以非极性氨基酸结尾(A-丙氨酸、V-缬氨酸都是非极性氨基酸)。
SARS-CoV-2属于β谱系B支系(乙型冠状病毒属Sarbe亚属),MERS-CoV属于β谱系C支系(乙型冠状病毒属Merbe亚属)。虽然隶属同一谱系(属),但二者差异巨大,亲缘关系(进化关系)极远。SARS-CoV-2、MERS-CoV二者Spike蛋白aa序列76%可比对,可比对部分一致度仅为34.91%。两种病毒的全基因组核苷酸序列仅28%可比对,可比对部分一致度为67.06%。
MERS-CoV发现于中东。为什么SARS-CoV-2 S1/S2位点左侧的四氨基酸序列与所有“近亲”大相径庭,却反而与基因组差距巨大,演化关系极远、发现地远隔重洋、相距万里的MERS-CoV如此相似?这种反常源自自然变异、自然演化,还是人为借鉴、人为赋予?
SARS-CoV-2、MERS-CoV间的结构关联、结构巧合还远不止于此。
前面提到,长furin切割序列的八个氨基酸位点从左对右通常依次用P4P3P2P1P1'P2'P3'P4'表示。SARS-CoV-2基本furin切割序列RRAR的四个氨基酸从左到右依次对应P4P3P2P1四个位点,RRAR左侧的第一个氨基酸位点称为P5位点。SARS-CoV-2、MERS-CoV P5位点的氨基酸皆为脯氨酸,即SARS-CoV-2基本furin切割序列RRAR、MERS-CoV基本furin切割序列RSVR左侧中的第一个氨基酸皆为脯氨基酸(P)。如下图所示:

SARS-CoV-2 、MERS-CoV furin切割序列P5位点皆为脯氨酸
PRRA、PRSV中的P表示脯氨酸,P1、P2、P3、P4、P5中的P则表示位点(position),二者含义不同。
SARS-CoV-2、MERS-CoV P5位点皆为脯氨酸,这是极不寻常的。furin切割序列P5位点极少出现脯氨酸,Thomas Lemmin等人发表的以下论文指出,在已识别的132个furin切割序列中,仅有5个序列P5位点为脯氨酸。
Structures and dynamics of the novel S1_S2 protease cleavage site loop of the SARS-CoV-2 spike glycoprotein
SARS-CoV-2刺突糖蛋白新型S1_S2蛋白酶切割位点环的结构和动力学
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7534663/
论文原文:
it
appears that a proline residue at position P5, i.e., the 5th residue
prior to the furin cleavage site, is rare and appears in only 5 out of
132 sequences (three mammalian and two viral sequences).
5个P5位点为脯氨酸的furin切割序列3个是哺乳动物序列,2个是病毒序列,2个病毒序列分别属于SARS-CoV-2、MERS-CoV。也就是说,furin切割序列P5位点为脯氨酸的病毒仅有两种,这两种病毒恰恰就是2012年出现的MERS-CoV和2019年出现的SARS-CoV-2。这实在是太过于巧合、太过于幽默了。
SARS-CoV出现于2002年,MERS-CoV出现于2012年,比之2022年,SARS-CoV-2的出现时间提前了约3年。
为什么SARS-CoV-2、MERS-CoV二者furin切割序列的P5位点都要使用脯氨酸呢?这是一个颇有深意的设计,P5位点的脯氨酸可能有以下多重作用:
1)、脯氨酸有特殊的构象刚性,可将furin切割序列对应的多肽与Spike蛋白的其他结构元件分开,使前者更好地暴露于furin等蛋白酶;
2)、脯氨酸产生的转角预计(可能未得到广泛确认)会导致PRRA插入两侧的2个丝氨酸(S673、S686)、1个苏氨基酸(T678 )形成O-连接聚糖(O-linked glycans),O-Linked聚糖能与人体细胞表面的某些蛋白质相互作用,形成一个可逃避免疫打击的糖链屏障,是SARS-CoV-2众多可能的免疫逃避/免疫屏蔽途径之一。
1)、2)、两点都是上面刚刚提到的Thomas Lemmin等人的论文所指出的。
3)、P5位点的脯氨酸还可能有助于调节Spike蛋白及病毒包膜的融合前构象,降低膜融合的能量门槛,使病毒包膜与细胞膜更容易发生膜融合,使病毒的感染(即细胞进入)更便捷、高效。这一推测基于英法两国科学家1 December 1998发表的以下论文:
A
Proline-Rich Motif Downstream of the Receptor Binding Domain Modulates
Conformation and Fusogenicity of Murine Retroviral Envelopes
受体结合域下游富含脯氨酸的基序调节鼠逆转录病毒包膜的构象和融合性
https://journals.asm.org/doi/10.1128/jvi.72.12.9955-9965.1998
4)、与丝氨酸(S)或苏氨酸(T)组成磷酸化基序/位点。
SARS-CoV-2 RRAR之前P5位点的脯氨酸P和P6位点的丝氨酸S组成的双氨基酸短序列SP恰好是脯氨酸定向激酶(proline-directed kinases)的两个作用基序之一;而MERS-CoV的对应位点恰好使用了脯氨酸定向激酶的另一个作用基序TP(苏氨酸-脯氨酸)。不得不叹服,远隔重洋、相距万里的SARS-CoV-2、MERS-CoV是何等的心心相印、心有灵犀,何等的默契!

MERS-CoV 、SARS-CoV-2 S1/S2位点左侧的的脯氨酸定向激酶作用基序TP、SP
SARS-CoV-2长furin切割序列RRAR'SVAS中的第2至第5氨基酸RARS还恰好符合嗜碱性激酶(basophilic kinases)发挥作用的RXXS 基序模式,SP及RARS中的两个丝氨酸S可分别被脯氨酸定向激酶、嗜碱性激酶有效磷酸化。磷酸化(Phosphorylation),指在蛋白质中的丝氨酸/苏氨酸/酪氨酸残基上加入一个磷酸基团。
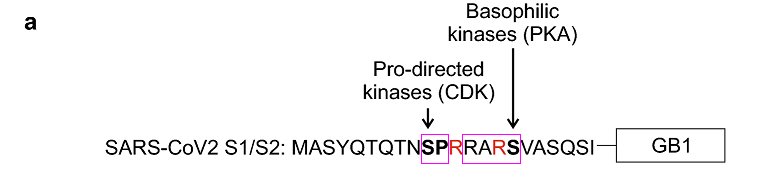
SARS-CoV-2 S1/S2位点附近的的脯氨酸定向激酶作用基序SP及嗜碱性激酶作用基序RARS
论文依据:
The sequence at Spike S1_S2 site enables cleavage by furin and phospho-regulation in SARS-CoV2 but not in SARS-CoV1 or MERS-CoV
https://www.nature.com/articles/s41598-020-74101-0
SARS-CoV-2、MERS-CoV间惊天地、泣鬼神的种种结构关联、结构巧合,是无目的、随机性自然变异、自然演化的一再奇迹,还是人为设计的蓄意而为、一脉相承?
SARS-CoV-2 Spike蛋白上存在十几个磷酸化位点(phosphorylation sites),其N、M、S、nsp3a、nsp9b等蛋白中共有约 70个磷酸化位点;SARS-CoV-2感染会干预、改变人体自身的磷酸化机制,导致宿主蛋白显著、严重的磷酸化改变,并显著增加某些生理过程(如碳代谢,carbon metabolism)中的磷酸化,SARS-CoV-2感染会导致人体增加2000 多种磷酸肽,在SARS-CoV-2感染的细胞中观察到超过15,000个宿主磷酸化位点。SARS-CoV-2感染还会改变(上调或下调)参与磷酸化的多种激酶的活性,并通过磷酸化与信号传导和细胞周期有关的蛋白质来干预、劫持宿主信号通路、信号传导,阻滞/停滞细胞周期,使细胞处于有利于病毒复制、病毒生存的周期阶段。
参考论文:
Frontiers _ SARS-CoV-2 Infection Triggers Phosphorylation Potential Target for Anti-COVID-19 Therapeutics.htm
https://www.frontiersin.org/articles/10.3389/fimmu.2022.829474/full
蛋白质磷酸化异常,包括磷酸化途径的改变会导致多种严重疾病,尤其是癌症。靶向磷酸化途径的药物代表了癌症治疗的有希望的领域。SARS-CoV-2有高度致癌性,SARS-CoV-2的致癌途径非常丰富。其它与磷酸化异常有关的疾病包括糖尿病、心血管疾病、阿尔茨海默症(Alzheimer's
disease)、炎症性疾病等等。
第五重借鉴:借鉴两种人类蛋白的PRRARS序列
SARS-CoV-2 S1/S2位点附近的PRRARS序列可能借鉴自人类Hermansky-Pudlak syndrome protein或某些RhoGEF蛋白。

SARS-CoV-2 S1/S2位点左侧的的PRRA序列及其延长序列PRRARS。
SARS-CoV-2 S1/S2位点左侧的的四氨基酸序列PRRA可能在所有其它已知病毒中都不存在。很多论文做过生物数据库搜索匹配,但未见有论文报告,何种其它病毒中的某种蛋白也含有PRRA序列;无人报告其它病毒具有PRRA序列,但下述论文却报告,PRRA序列及其延长序列-六氨基酸序列PRRARS序列存在于两种人类蛋白之中。
论文依据:
The polybasic insert, the RBD of the SARS-CoV-2 spike protein, and the feline coronavirus – evolved or yet to evolve
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7833556/
论文原文:
We
scanned the human databases (Taxon 9606) and found that the exact
sequence PRRARS was present only in two human proteins: AAB17869.1,
Hermansky-Pudlak syndrome protein (HPS1), and AAF79955.1, RhoGEF. There
are 17 isoforms of HPS1; incidentally, RhoGEF in the curated UniProt
data does not carry this motif (Supplemental Table 1).
两种含有PRRARS序列的人类蛋白是HPS1 (Hermansky-Pudlak syndrome protein 1)和RhoGEF,某些生物库(如UniProt )中的RhoGEF蛋白不包含PRRARS序列。
Hermansky-Pudlak syndrome(赫曼斯基-普德拉克综合症,HPS)是一类遗传性的综合眼皮肤白化病,对应六种基因突变,HPS1是其中一种突变蛋白。患有Hermansky-Pudlak syndrome的人皮肤容易损伤,容易皮肤淤紫、鼻出血、齿龈出血、咯血、手术后出血及产后出血,容易患皮肤癌。
RhoGEF (Rho Guanine Nucleotide Exchange Factor,Rho 鸟嘌呤核苷酸交换因子) 直接负责响应多种细胞外刺激而激活Rho家族GTP酶,在调节多种细胞反应(例如增殖、分化和运动)及基本细胞过程(例如细胞形态和极性,细胞周期进程和基因转录)中起关键作用。
论文指出,某些生物库(如UniProt )中的RhoGEF蛋白不包含PRRARS序列,这或许意味着,PRRARS序列只存在于某些RhoGEF蛋白中,不清楚包含PRRARS序列的RhoGEF蛋白是否也是变异蛋白。
号称来处动物的SARS-CoV-2具有的,其它病毒无一具有的,与人类ENaC-α相同的长furin切割序列RRAR'SVAS是如何得来的?SARS-CoV-2具有的,其它病毒同样无一具有的,存在于人类变异蛋白--Hermansky-Pudlak syndrome protein中的PRRARS序列又是如何得来的?是SARS-CoV-2在动物体内自然变异、自然演化产生的,还是人为借鉴、人为赋予的?
(未完待续)

















