接續:連繫新冠病毒與Ralph S. Baric的雙紐帶(中)
新冠病毒決定ACE2結合能力,決定細胞進入(感染)能力,決定宿主範圍的五個RBD關鍵氨基酸是人為設計的,有人參照WIV1病毒的關鍵氨基酸,通過氨基酸重用和等價、等效替代,設計出了新冠病毒的關鍵氨基酸。人為設計的新冠病毒的關鍵氨基酸,與WIV1(rs3367)病毒的關鍵氨基酸高度等價、等效(三個相同,一個高度等價,整體應該高度同構,與ACE2結合高度等效),這兩個病毒關鍵氨基酸的高度關聯,高度等價、等效性是以下三項“巧合”的內在原因:
1)新冠病毒、WIV1(rs3367)病毒的跨物種感染能力都極為強大,難分伯仲。
2)新冠病毒、WIV1(rs3367)病毒的宿主範圍都極為廣泛,而且,它們的宿主範圍高度雷同,或者說,它們能感染的物種高度重合。
3)新冠病毒、WIV1(rs3367)病毒的刺突蛋白都極為適宜結合hACE2(human ACE2),它們的刺突蛋白與hACE2的結合親和力都極為強大。
本系列上篇文章已經詳細介紹、說明了“巧合”1)、2),第3)項“巧合”的依據如下:
a)本系列中篇文章中的論文A-2(Ralph S. Baric團隊2016年3月14日的PNAS論文)以小鼠體內實驗證明:WIV1病毒刺突蛋白與hACE2的結合親和力遠強於它與mACE2的結合親和力,WIV1-CoV(WIV1的實驗室克隆)及WIV1-MA15(刺突蛋白來自WIV1的實驗室嵌合病毒)兩種病毒在表達hACE2的轉基因小鼠(mACE2轉基因為hACE2的小鼠)肺中的複製滴度,比它們在普通小鼠肺中的複製滴度高100倍!這兩種病毒只能使普通小鼠出現輕微臨床症狀,但它們能使表達hACE2的轉基因小鼠體重減輕超過10%;它們在轉基因小鼠大腦中的複製比在肺中更強勁,使部分轉基因小鼠患上了致命性腦炎。相關內容可參見中篇文章論文A-2要點7)。
b)澳大利亞弗林德斯大學的論文:新冠刺突蛋白與ACE2結合親和力的跨物種模擬比較及其對病毒起源的指示(2020年5月13日發表於預印本平台arxiv,2021年6月24日正式發表於Nature Science),通過分子動力學定量模擬研究,計算出了新冠S蛋白(刺突蛋白)與各物種ACE2相互作用的結合能(binding energies for the interactions),各物種ACE2與新冠S蛋白的結合能從高到低依次為:人類 > 穿山甲 > 狗 > 猴子 > 倉鼠 > 雪貂 > 貓 > 老虎 > 蝙蝠 > 麝貓 > 馬 > 牛 > 蛇 > 老鼠。定量模擬計算結果表明:在14個對照研究物種中,與新冠病毒刺突蛋白結合能力最強的物種ACE2,是hACE2!
https://www.nature.com/articles/s41598-021-92388-5
其實還有第四項“巧合”:新冠病毒、WIV1病毒都具有泛組織、器官感染能力。這當然首先與ACE2的泛組織、器官存在性有關;它應該同時與新冠RBD關鍵氨基酸的人為設計有關,因為這一人為設計賦予了新冠病毒強大的hACE2結合能力,使它能夠非常有力、高效地結合人體各個組織、器官細胞的跨膜糖蛋白受體--ACE2,非常有力、高效地感染人體各個組織、器官(的細胞)。
是誰參照WIV1(rs3367)病毒的關鍵氨基酸,通過復用和等價、等效替代,設計出了新冠病毒的關鍵氨基酸?
設計出新冠病毒關鍵氨基酸的人,肯定對WIV1(rs3367)病毒,對關鍵氨基酸,都做過深入研究。
有一個病毒學家,及他領導的科學團隊,不僅對WIV1(rs3367)病毒、對冠狀病毒的RBD關鍵氨基酸都做過深入的研究,而且,還同時滿足以下五個條件:
1)反覆、深入研究過WIV1(rs3367)病毒;
2)“無數次”研究過冠狀病毒的RBD(Receptor Binding Domain,受體結合域)、RBD的關鍵氨基酸,對它們與ACE2,特別是與hACE2的作用機制瞭如指掌;
3)有着長期的,極其豐富的冠狀病毒改造、合成經驗,功能增益研究經驗,無數次人工合成自然冠狀病毒(克隆)、常規改造冠狀病毒、功能增益性改造冠狀病毒;
4)多次實施RBD關鍵氨基酸的改造、替代實驗,研究它們的改變對物種感染能力,包括對人體細胞感染能力的影響,改造、培育出過多種可跨物種傳播的冠狀病毒,如可感染人類和靈長類動物的冠狀病毒;
5)曾以WIV1(rs3367)病毒為原材料進行功能增益性的冠狀病毒嵌合實驗,改造出了有人類致病能力的危險病毒WIV1-MA15。
這個病毒學家是誰?就是冠狀病毒研究的全球頂級權威Ralph S. Baric。Ralph S. Baric及其團隊應該是唯一全部符合以上五個條件的個人、團隊。
Ralph S. Baric以模擬、預測病毒自然突變的名義對冠狀病毒進行功能增益研究,其眾多論文的實質就是在探索、嘗試:
如何將不能感染人類的動物來源的冠狀病毒改造為可感染人類的冠狀病毒,
如何將沒有人類致病力的動物來源的冠狀病毒改造為可使人類致病的冠狀病毒,
如何增強、完善實驗室冠狀病毒對人類及其它動物的感染、致病能力。
Ralph S. Baric(團隊)對關鍵氨基酸的研究
Ralph S. Baric團隊不僅對雙紐帶之一--WIV1(rs3367)做過反覆、深入的研究(中篇文章已經介紹了Ralph S. Baric團隊研究WIV1、rs3367的四篇論文),而且對另一紐帶--冠狀病毒的RBD關鍵氨基酸也做過極其頻繁、極其細緻、極其深入、極其透徹的研究。
Ralph S. Baric幾乎每一篇論文都要研究冠狀病毒的刺突蛋白,研究刺突蛋白的RBD,其研究RBD關鍵氨基酸的論文非常之多,本文將提供四篇示例。其中,論文B-1、B-2就是中篇文章中的論文A-1、A-2,Ralph S. Baric團隊在這二篇論文中同時研究了WIV1病毒和冠狀病毒的RBD關鍵氨基酸。
論文B-1
2015年11月9日,Ralph S. Baric團隊在《自然醫學》(Nature Medicine)雜誌發表了著名的嵌合病毒論文:A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence(一個類似SARS的蝙蝠冠狀病毒群顯示了產生人類流行疫情的潛力)
https://www.nature.com/articles/nm.3985
該論文就是中篇文章中的論文A-1。WIV1在這篇論文中是病毒主角SHC014的對照病毒。
論文分別比較了WIV1與SARS,SHC014與SARS兩對病毒RBD關鍵氨基酸的異同。論文指出:在刺突蛋白決定ACE2結合能力、細胞進入能力及宿主範圍的5個RBD關鍵氨基酸(殘基)中,WIV1病毒與SARS病毒雖然有三個關鍵氨基酸(1、2、4)不同,但其刺突蛋白仍能與hACE2(human ACE2)有效結合。
WIV1、SARS關鍵氨基酸對照表。二者的第3、5關鍵氨基酸相同,第1、2、4關鍵氨基酸不同。
WIV1是具有人體細胞進入(感染)能力的特殊蝙蝠冠狀病毒,其刺突蛋白可有效結合hACE2,這是石正麗團隊2013年10月30日的nature論文已得出的結論,前面的描述引用了這一結論。
https://www.nature.com/articles/nature12711
論文接着指出,相比WIV1,SHC014與SARS關鍵氨基酸差異更大,二者的關鍵氨基酸無一相同。
SHC014、SARS關鍵氨基酸對照表,它們的5個關鍵氨基酸無一相同。
SHC014的RBD關鍵氨基酸與SARS迥異,它的刺突蛋白能否如SARS的刺突蛋白那樣,有效結合hACE2呢?它能否如SARS那樣,進入(感染)人體細胞呢?這是該論文要研究的核心問題之一(hACE2結合能力與人體細胞進入能力是等價的)。
為解決上述問題,Baric團隊用SHC014的刺突蛋白和SARS-CoV-MA15的骨架構建了嵌合病毒SHC014-MA15,並實施了該病毒對人氣道上皮的感染實驗。實驗結果表明,SHC014-MA15能有效感染人氣道上皮組織,並在組織細胞內大量複製。這即是證明了:SHC014的刺突蛋白也能有效結合hACE2,SHC014與WIV1一樣,也是具有人體細胞進入(感染)能力的特殊蝙蝠冠狀病毒。
註:WIV1、SHC014都有人體細胞進入(感染)能力,同時又都沒有人體致病能力,他們都不會使人體產生疾病症狀;絕大多數蝙蝠冠狀病毒都沒有人體細胞進入(感染)能力,因而沒有人體致病能力。
所有已知的蝙蝠冠狀病毒,無論是否有人體細胞進入(感染)能力,它們統統都沒有人體致病能力,不存在已知的有人體致病能力的蝙蝠冠狀病毒(發現自蝙蝠的冠狀病毒)!
論文B-2
2016年3月14日,Ralph S. Baric團隊在PNAS(美國國家科學院院刊)發表了如下論文:SARS-like WIV1-CoV poised for human emergence(類SARS冠狀病毒WIV1-CoV對人類有潛在威脅)
https://www.pnas.org/content/113/11/3048
該論文就是中篇文章中的論文A-2。在這篇論文中,Ralph S. Baric團隊對比了SARS-WT(即SARS-CoV)、SARS-MA15、WIV1三種冠狀病毒與hACE2直接相互作用的14個RBD氨基酸殘基,包括五個關鍵氨基酸殘基,見圖A;同時深入、細緻地對照研究了SARS-CoV、WIV1這兩種病毒的刺突蛋白與hACE2的接觸、結合模型,特別是442、472、487三個RBD關鍵氨基酸殘基與hACE2的作用情況,見圖B。
為便於大家理解,我對圖A、圖B再作一些補充說明。
SARS-WT,SARS-CoV wild type,指SARS病毒的野生毒株;SARS-MA15,即SARS-CoV-MA15,Ralph S. Baric團隊在實驗室中培育的SARS-CoV的小鼠適應性變異體,SARS-CoV能感染小鼠細胞,但不會使小鼠產生臨床症狀,SARS-CoV-MA15則可使小鼠嚴重致病並致死。
圖A是SARS-WT、SARS-MA15、WIV1-CoV三種病毒刺突蛋白RBD與hACE2直接接觸的14個氨基酸殘基,後二種病毒與SARS-WT不同的氨基酸殘基分別以深紅或深藍背景色突出顯示,紫紅色長條框標出了它們的5個RBD關鍵氨基酸殘基。
SARS病毒的五個關鍵氨基酸(殘基)依次是刺突蛋白的第442、472、479、487、491氨基酸(殘基),
WIV1病毒的五個關鍵氨基酸(殘基)依次是刺突蛋白的第443、473、480、488、492氨基酸(殘基)。

WIV1、SARS關鍵氨基酸對照表,它們的第442、472、487三個關鍵氨基酸殘基不同。
圖A中14個氨基酸的序號基於SARS,WIV1的氨基酸都向前移了一位,以與SARS的對應氨基酸對齊。
圖B左圖是SARS病毒刺突蛋白S1亞基(也叫受體結合亞基,S1亞基前端為RBD)與hACE2的作用模型,圖B右圖是WIV1病毒刺突蛋白S1亞基與hACE2的作用模型。
圖B兩子圖中,右側的雙螺旋結構是hACE2,其上的橙黃色部分是hACE2自身的接觸殘基;
圖B兩子圖中,左側是兩種病毒刺突蛋白的S1亞基,SARS的S1亞基以灰黑色表示,WIV1的S1亞基以藍色表示。紅色圖案代表兩病毒相同的RBD氨基酸接觸殘基,圓圈處是二者三個不同的關鍵氨基酸殘基(442、472、487)與hACE2殘基的作用情況。
論文B-3
2007年12月19日,Ralph S. Baric團隊於Journal of Virology(病毒學雜誌)在線發表了一篇論文:Mechanisms of Zoonotic Severe Acute Respiratory Syndrome Coronavirus Host Range Expansion in Human Airway Epithelium(人畜共患SARS冠狀病毒在人氣道上皮中擴大宿主範圍的機制)
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2258931/
https://journals.asm.org/doi/10.1128/JVI.02041-07

Ralph S. Baric仍是論文的通訊作者。
這篇論文主要做了兩件事:
1)演示了如何對病毒的關鍵氨進行人為替換,如何在人氣道上皮中培養、篩選病毒,如何將不能感染人類的動物來源的冠狀病毒簡易、快捷地改造、培育為可感染人氣道上皮的,可跨物種感染、傳播的冠狀病毒。
具體而言,是對果子狸冠狀病毒SZ16的第三關鍵氨基酸進行了人為替換,並將中間病毒放到人氣道上皮中培養、篩選,成功改造、培育出了兩種可感染人類和靈長類動物的冠狀病毒。
2)極為深入地研究、解讀了關鍵氨基酸與hACE2的作用機制,以及關鍵氨基酸替代、改變對ACE2結合能力、細胞感染能力的影響機理。
論文病毒改造的原材料是果子狸(civet,有時譯作麝香貓)類SARS冠狀病毒SZ16(SARS-CoV SZ16),該病毒分離自SARS疫情期間中國廣東地區動物市場中的果子狸。SZ16的刺突蛋白與SARS流行病毒株SARS-Urbani的刺突蛋白有18個氨基酸差異,其中16個位於S1亞基(S1亞基是受體結合亞基,S2亞基是膜融合亞基,受體結合域RBD是S1亞基的一部分)。
論文列出了SZ16、SARS-Urbani、SARS-GD03三種病毒株刺突蛋白的氨基酸差異,見下圖:
藍色表示動物(果子狸)來源的氨基酸,黃色表示SARS疫情早期出現的氨基酸,橙棕色表示疫情中期出現的氨基酸,紅棕色表示疫情晚期出現的氨基酸。
SARS-Urbani是SARS疫情晚期分離自美國的一個SARS病毒株;
GD03是比Urbani更晚出現的SARS病毒株,它是2004年1月疫情再次零星出現時,分離自一散發廣東患者的SARS病毒株。由圖可見,它的部分氨基酸似乎出現了返祖現象。
SARS疫情發生於2002年11月16日-2003年9月2日,高峰期為2002年11月16日-2003年7月16日。
研究者首先用SZ16刺突蛋白的基因序列,以及SARS-Urbani刺突蛋白以外部分的基因序列,設計了一個嵌合的基因序列,而後基於嵌合的基因序列,使用反向遺傳平台合成、收穫了嵌合病毒icSZ16-S(基於基因序列合成病毒,這是反向遺傳平台的基本功能)。實驗證明,icSZ16-S不能感染Vero E6細胞(非洲綠猴腎細胞系細胞),也不能感染HAE細胞(人氣道上皮細胞)。
註:
ic:infectious cDNA,ic前綴表示使用反向遺傳平台合成的有生物活性的冠狀病毒。SZ16-S表示嵌合病毒icSZ16-S的S蛋白來自SZ16(骨架來自SARS),S蛋白即Spike蛋白,就是刺突蛋白。
HAE細胞:human airway epithelium cells,人氣道上皮細胞,包括纖毛上皮細胞、非纖毛上皮細胞以及杯狀細胞。
接下來,研究者對icSZ16-S的一個RBD關鍵氨基酸進行了替換,人為製造了一個氨基酸突變K479N:
即將icSZ16-S刺突蛋白的第479氨基酸--賴氨酸K(來自SZ16),替換為SARS刺突蛋白的第479氨基酸--天冬酰胺N(亦稱為天門冬酰胺)。第479氨基酸是SARS及SZ16五個RBD關鍵氨基酸中的第三個。
SZ16的第479氨基酸是賴氨酸K,SARS-Urbani的第479氨基酸是天冬酰胺N。
基於K479N替換後得到的基因序列,再次使用反向遺傳平台,合成、收穫了第二個實驗室病毒:icSZ16-S 479N。相比不能感染Vero E6細胞的icSZ16-S,icSZ16-S 479N有了一定的“進步”:實驗證明,icSZ16-S 479N能感染Vero E6細胞,並能在細胞中低水平地複製,衰弱地傳代、傳播;icSZ16-S 479N仍然不能感染HAE細胞。
為使icSZ16-S 479N適應HAE細胞,研究者將其放到人氣道上皮樣本中持續培養,並按一定的策略反覆進行傳代和優化篩選。
培養、傳代、篩選第8天,得到了第三個實驗室病毒icSZ16-S K479N D8。相比icSZ16-S K479N,D8在人氣道上皮組織中發生了一個適應性變異Y442F:刺突蛋白第442氨基酸由酪氨酸Y變異為苯丙氨酸F;
培養、傳代、篩選第22天,得到了第四個實驗室病毒icSZ16-S K479N D22。D22在Y442F變異的基礎上,又發生了另一個適應性變異L472F:刺突蛋白第472氨基酸由亮氨酸L變異為苯丙氨酸F。
SARS、SZ16刺突蛋白的第442氨基酸就是它們的第一個RBD關鍵氨基酸,第472氨基酸就是它們的第二個RBD關鍵氨基酸。也就是說,人為替換第479氨基酸(第三個RBD關鍵氨基酸)改造出的icSZ16-S 479N,在人氣道上皮環境中,先後發生了另外二個關鍵氨基酸的適應性突變,變異出兩種新病毒。
相關病毒關鍵氨基酸對照表,表中展示了K479N替換、Y442F變異、L472F變異所涉及的關鍵氨基酸
有一個小小的巧合,新冠病毒、WIV1病毒、icSZ16-S K479N D22病毒的第二個RBD關鍵氨基酸都是苯丙氨酸F。
S蛋白氨基酸序列長度為1269、1273的新冠、WIV1、SARS、D22五病毒關鍵氨基酸對照表
能感染人氣道上皮(HAE)細胞的病毒已經產生了。
實驗證明:
1)icSZ16-S K479N D22能有效感染HAE細胞、Vero E6細胞、DBT-hACE2細胞,並有效複製,但它在三種細胞中的複製能力或多或少弱於icSARS(使用反向遺傳平台合成的SARS流行毒株Urbani的克隆毒株);
2)icSZ16-S K479N D8也能感染上述三種細胞,但它在這些細胞中的複製能力或多或少弱於D22;
3)icSZ16-S K479N在HAE細胞和DBT-hACE2中的複製能力低下,複製滴度與D22、D8相差幾個數量級;
4)icSARS、icSZ16-S K479N、icSZ16-S K479N D8、icSZ16-S K479N D22都不能感染未轉基因的普通DBT細胞。
稍後提供的四病毒對四細胞的感染實驗數據圖將更形象、具體地展示以上實驗結果。
註:
DBT-hACE2是論文研究者設計、製造了一種表達hACE2的轉基因小鼠DBT細胞,其細胞的跨膜糖蛋白--ACE2受體被轉基因為hACE2。
DBT:Delayed Brain Tumor,延遲腦腫瘤;小鼠DBT細胞,即小鼠延遲腦腫瘤細胞,或稱為小鼠星形細胞瘤遲發性腦瘤細胞。
icSARS、K479N、D8、D22對HAE、Vero E6、DBT-hACE2、普通DBT細胞的感染實驗數據圖。
橫坐標:感染後時間,單位為小時;縱坐標:細胞培養物中的病毒滴度,單位為PFU/ml。
註:PFU,Plaque-forming unit,單位體積的病毒形成的空斑數,是代表有活性的病毒粒子濃度的單位。空斑(Plaque)是病毒誘導宿主細胞裂解形成的點狀物,可通過顯微鏡觀察。
為研究各病毒RBD及RBD關鍵氨基酸與hACE2的作用、結合機制,研究者使用蛋白質結構設計、預測工具Rosetta Design生成了SZ16、icSZ16-S K479N、icSZ16-S K479N D8和icSZ16-S K479N D22等病毒的RBD結構模型,並將這些結構模型疊加到SARS Urbani RBD與hACE2相互作用的晶體結構模型上,得到了以上病毒與hACE2相互作用的晶體結構模型。
基於RBD-hACE2相互作用的晶體結構模型,研究者極為深入地分析了各病毒442、472、479、487四個RBD關鍵氨基酸與hACE2的具體結合或作用情況(沒有涉及最保守的第五關鍵氨基酸,即刺突蛋白的第491氨基酸,這幾個病毒的第491氨基酸全都相同,都是酪氨酸Y),解釋了不同的RBD關鍵氨基酸,如何導致了不同的hACE2結合能力,不同的細胞進入能力。具體分析、解讀如下:
a)SZ16的第479氨基酸殘基--Lys479(賴氨酸K),似乎與hACE2的結合熱點殘基Lys31和His34存在靜電排斥(碰撞),因而其RBD不能與hACE2良好結合,見圖B。圖B中lys31和His34兩個hACE2殘基對應的點密集團塊為深褐色,表示這兩個hACE2殘基與附近的RBD殘基存在衝突(靜電排斥或碰撞)。
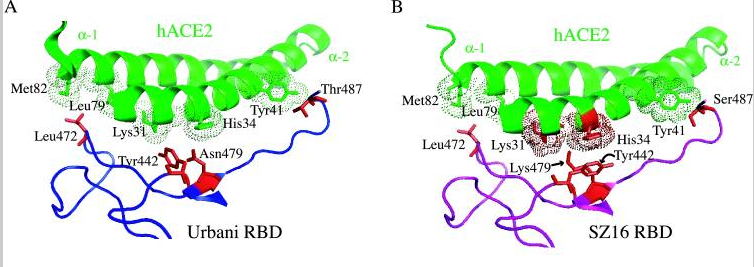
b)引入點突變K479N,即將479氨基酸替換為SARS Urbani 的479氨基酸--天冬酰胺N(Asn479)後,479位點與hACE2殘基的排斥力被消除,因而icSZ16-S K479N的刺突蛋白能夠與hACE2結合,見圖C。圖C中lys31和His34兩個hACE2殘基對應的團塊為淺綠色,表示它們與周圍的RBD殘基相互吻合。
c)SARS Urbani 442、479兩個RBD關鍵氨基酸殘基分別與兩個不同的hACE2殘基相互作用(殘基442與Lys31相互作用,殘基479與His34相互作用),見圖A,二對作用互不干擾,互不競爭,因而結合親和力較強;儘管icSZ16 K479N 的442、479殘基與Urbani相同,但這兩個殘基預計會同時與hACE2殘基Asp32作用,而且,殘基442預計還會同時與Lys31、Asp32這兩個hACE2殘基作用,見圖C。這種殘基作用的交叉競爭削弱了RBD與hACE2的結合親和力,因而,icSZ16 K479N的刺突蛋白雖然能與hACE2結合,但結合能力較弱。
d)icSZ16-S K479N、SARS Urbani唯一不同的RBD關鍵氨基酸是第487氨基酸,即第四個RBD關鍵氨基酸(參見前面的“相關病毒關鍵氨基酸對照表”)。前者的第487氨基酸是絲胺酸-S(Ser487),它只與一個hACE2殘基(Tyr41)結合,見圖C;SARS Urbani 第487氨基酸是蘇胺酸-T(Thr487),它獨自結合了三個hACE殘基,而且沒有其它RBD殘基交叉競爭與這三個hACE殘基的結合,見圖A(圖A似乎看不到與Thr487作用的三個hACE2殘基?)。Urbani的487殘基與hACE2的氫鍵強於K479N的487殘基與hACE2的氫鍵,這是Urbani與hACE2的結合親和力強於icSZ16-S K479N的另一個原因。
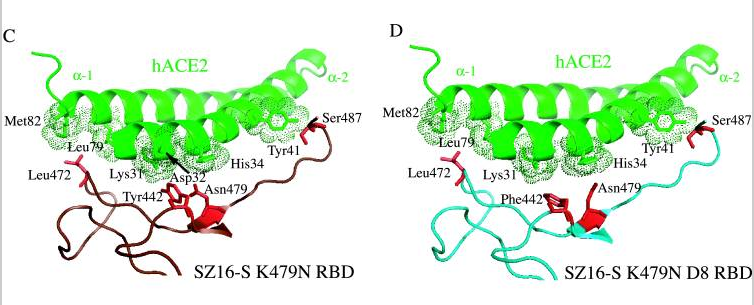
e)Rosetta Design模型預測icSZ16-S K479N D8的Y442F突變消除了K479N的442、472殘基對hACE2殘基的交叉競爭,D8的F442(即Phe442)、N479殘基(Asn479)各自與不同的hACE2殘基作用(F442結合Lys31,N479結合His34);同時,它的S487殘基(Ser487)與hACE2的Tyr41殘基之間的相互作用和氫鍵網絡保持不變,見圖D。這使得icSZ16-S K479N D8與hACE2的結合能力大大提高,因此,D8在HAE細胞和DBT-hACE2細胞中的生長(感染、複製)比K479N茁壯得多。
f)Rosetta Design模型預測icSZ16-S K479N D8的L472殘基(Leu472)無交叉競爭地同時結合了兩個hACE2殘基(Leu79、Met82),見圖D;icSZ16-S K479N D22由於發生了L472F突變(亮氨酸L突變為苯丙氨酸F),預計其F472殘基(Phe472)將無交叉競爭地同時結合三個hACE2殘基(Leu79、Met82、Tyr83),見圖E,Rosetta Design還預測F472殘基將與這三個hACE2殘基產生強烈的疏水相互作用,進一步加強彼此的結合。因為上述原因,L472F突變增強了RBD與hACE2的結合親和力,所以,D22在HAE細胞和DBT-hACE2細胞中比D8更具生長優勢。
以上各圖中,hACE2邊緣的點密集團塊表示與RBD氨基酸殘基相距不超過4Å(1Å=0.1納米)的hACE2殘基,預計它們會與鄰近的紅色圖案表示的RBD氨基酸殘基發生相互作用。
在論文前面“材料和方法”中的“SARS冠狀病毒尖峰的系統發育分析和計算機建模”小節,論文仿照479替換衍生442、476突變這一模式,提出了如下RBD氨基酸替換研究模式:利用Rosetta Design web server及分子建模工具MacPyMol,對SARS病毒RBD既有氨基酸的各種替換、突變可能性進行模擬,為每個位點氨基酸的替換、改變生成十個其它位點衍生替換、改變的模型,為每個模型再生成十個次級衍生替換、改變模型,如此反覆迭代,通過比較前後生成的各模型與hACE2的結合能,從中選出與hACE2結合的最佳關鍵氨基酸模型或最佳RBD氨基酸模型。
(這段內容在論文的後續部分似乎沒有展開)
如果我的理解沒有錯的話,通過上述建模和算法,Ralph S. Baric團隊能得出與hACE2結合的冠狀病毒最佳RBD關鍵氨基酸組合甚至最佳RBD氨基酸序列,所得結果可能被用於設計或輔助設計新冠病毒關鍵氨基酸,及新冠病毒RBD氨基酸。
在論文Disscussion部分,研究者意猶未盡地寫道:
顯然,可能存在多種遺傳途徑來允許人畜共患病SARS冠狀病毒宿主範圍的擴大,確定其他接觸界面點突變是否能增強人畜共患病病毒對HAE細胞(人氣道上皮細胞)的感染將是一件有趣的事情。
論文B-3也是一篇SARS相關的功能增益研究論文,它改變了冠狀病毒的感染特性和宿主範圍,它通過關鍵氨基酸的人為替換和人為創造的人氣道上皮培養環境,用沒有人類、靈長類感染能力的果子狸冠狀病毒SZ16改造、培育出了可以感染人氣道上皮細胞(HAE)和靈長類動物Vero E6細胞的可跨物種感染、傳播的冠狀病毒icSZ16-S K479N D8和icSZ16-S K479N D22。
D8和D22還能感染BALB/c小鼠(白變種實驗室小鼠)的肺細胞,並在小鼠肺中良好生長、複製,不過,它們都不會使BALB/c小鼠產生臨床症狀。
論文B-4
2008年9月1日(Published online 2008 Jun 25),Ralph S. Baric等人在Journal of Virology發表了論文:Pathways of Cross-Species Transmission of Synthetically Reconstructed Zoonotic Severe Acute Respiratory Syndrome Coronavirus(人畜共患嚴重急性呼吸綜合徵冠狀病毒的跨物種傳播途徑)
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2519660/
https://journals.asm.org/doi/10.1128/JVI.00818-08
Ralph S. Baric依然是論文的通訊作者。

論文B-4是論文B-3研究的延續。
論文研究了7種病毒對不同細胞的感染能力,它們是:
病毒1:嵌合病毒icSZ16-S,它的刺突蛋白來自果子狸冠狀病毒SZ16,骨架來自SARS病毒株Urbani,
病毒2:icSZ16-S K479N,它是對icSZ16-S的479氨基酸實施人為替換(K479N)後得到的病毒,
病毒3:icSZ16-S K479N D22,它是icSZ16-S K479N在人氣道上皮上培養、傳代、篩選得到的實驗室變異體,
病毒4-6:三個SARS病毒的克隆毒株:icSARS、icCUHK-W1、icGZ02,
病毒7:SARS不同毒株的人工混嵌體icGD03-S,它應該是SARS-GD03的刺突蛋白與SARS-Urbani的骨架嵌合而成的。
關於病毒4-6的更多說明:
icSARS是用反向遺傳平台合成的SARS-Urbani的克隆,SARS-Urbani是SARS疫情晚期分離自美國的SARS病毒株。
GZ02是SARS疫情早期分離自廣州的SARS病毒株。
CUHK-W1是SARS疫情中期分離自香港的SARS病毒株。
GD03是比Urbani更晚的SARS病毒株,它是2004年1月疫情再次零星出現時分離自一散發廣東患者的SARS病毒株。
(SARS疫情發生於2002年11月16日-2003年9月2日,高峰期為2002年11月16日-2003年7月16日)
論文詳盡地列出了所涉及各病毒刺突蛋白的氨基酸差異,並標出了其它6種刺突蛋白相對於SARS-Urbani刺突蛋白的氨基酸差異數,見圖D;
論文還細緻地列出了cACE2(civet ACE2)及hACE2或primate-ACE2(靈長類動物ACE2)的所有熱點氨基酸殘基及與它們發生作用或接觸的SARS-Urbani的RBD氨基酸殘基,見圖E。
前一論文(B-3)研究、實驗了相關病毒對HAE細胞(人氣道上皮細胞)、Vero E6細胞、普通小鼠DBT細胞、轉基因小鼠DBT-hACE2細胞的感染複製情況。論文B-4又引入了另一種轉基因小鼠DBT細胞:DBT-cACE2,以進一步研究相關病毒的感染、複製能力,對照分析它們的RBD、關鍵氨基酸與不同ACE2的作用機制。
DBT-cACE2細胞是將普通小鼠DBT細胞中的ACE2轉基因為cACE2(civet ACE2,果子狸或麝香貓的ACE2)而得到的。
相關實驗數據見下圖。
7種病毒對4種細胞中的感染、複製實驗數據圖。
橫坐標是感染後時段,單位為小時;縱坐標是病毒的複製滴度,單位為PFU/ml。
以上實驗數據圖表明:
a)icSZ16-S K479N、icSZ16-S K479N D22及icCUHK-W1不能在DBT-cACE2細胞中有效複製,或只能低水平複製,而其它病毒,包括icSZ16-S都能在DBT-cACE2細胞中有效複製,見圖A;
b)icSZ16-S不能在DBT-hACE2細胞中有效複製,而其它病毒都能在DBT-hACE2細胞中不同程度地複製(K479N複製能力相對較差),見圖B;
c)icSZ16-S不能在Vero E6細胞中有效複製,而其它病毒都能在Vero E6細胞中不同程度地複製,GZ02、CUHK-W1、icSARS、icSZ16-S K479N D22在Vero E6細胞中的複製都極為強烈,見圖C;
(論文還指出:上述七種病毒皆不能感染未轉基因的普通小鼠DBT細胞。)
可見,icSZ16-S與icSZ16-S K479N及icSZ16-S K479N D22在細胞感染、複製能力,物種ACE2結合能力上存在巨大差異。
icSZ16-S、icSZ16-S K479N二者的刺突蛋白只有一個氨基酸不同,即第479氨基酸,icSZ16-S的第479氨基酸是賴氨酸K,而icSZ16-S K479N的第479氨基酸被人為替換為SARS-Urbani的第479氨基酸--天冬酰胺N(人為製造了K479N突變)。刺突蛋白第479氨基酸是第三個RBD關鍵氨基酸。
icSZ16-S、icSZ16-S K479N D22二者的刺突蛋白有三個氨基酸不同,除了K479N差異外,它們還有另外兩個氨基酸差異:Y442F和L472F。刺突蛋白第442、472氨基酸分別是第一、第二個RBD關鍵氨基酸。

相關病毒RBD關鍵氨基酸對照表
icSZ16-S、K479N、K479N D22這三個病毒在不同物種ACE2結合能力,不同物種細胞感染、複製能力上的巨大差異就是它們RBD關鍵氨基酸的差異造成的。
為了解釋各病毒與cACE2、hACE2的不同結合能力,研究者使用RosettaDesign和Modeler軟件開發了4種病毒(SARS-Urbani、SZ16、icSZ16-K479N、SZ16-K479N D22)刺突蛋白與cACE2、hACE2相互作用的結構模型,深入分析了各病毒與cACE2、hACE2在各個關鍵氨基酸位點的作用情況,解讀了關鍵氨基酸差異對hACE2結合能力的影響。論文指出:
1)cACE2分子與hACE2分子有兩個關鍵殘基(E30和Y34)不同,cACE2的E30和Y34的附加甲基可能會使cACE2結合界面產生明顯的突起。SARS-Urbani的RBD能夠良好適應hACE2的結合界面,見圖A;同時,它又能容納cACE2的表面突起,見圖B。因此,SARS-Urbani對人類和civet(果子狸或麝香貓)有雙重宿主適應能力;
2)與Urbani RBD類似,SZ16 RBD也能容納cACE2的E30、Y34造成的表面突起,以實現有效結合,見圖C;
3)SZ16 K479N中的K479N突變(人為替換)引入的479N(天冬酰胺)重塑了SZ16刺突蛋白的結合界面,促進了其與hACE2的結合,見圖D;
4)見圖E,479N對SZ16 K479N結合界面的重塑使RBD氨基酸殘基(V404、Y440)與cACE2氨基酸殘基(E30、Y34)之間產生衝突(綠色表示吻合,褐色表示存在衝突,深褐色表示衝突難以克服的區域),SZ16 K479N RBD無法容納cACE2的延長突出部分,這阻礙了二者的結合。因此,K479N突變(人為替換)使RBD獲得了結合hACE2的能力,但同時失去了結合cACE2的能力;
5)在K479N基礎上,Y442F和L472F突變進一步重塑了SZ16 K479N D22的RBD,進一步提高了RBD與hACE2的結合效率,見圖F;
6)與SZ16 K479N一樣,SZ16 K479N D22的RBD也不能容納cACE2的延長突出部分,從而使二者無法結合,見圖G。
和論文B-3一樣,論文B-4也是功能增益研究論文。本文介紹的四篇論文全都是SARS相關的功能增益研究論文。這四篇論文無一例外,全都得到了美國國立衛生研究院(NIH)下屬的美國國家過敏和傳染病研究所(NIAID)的資助、支持。十幾年來,NIAID的所長,一直是現白宮首席醫學顧問,聲稱新冠病毒來自自然界的福奇。
由論文B-3、B-4可知:
早在2007、2008年,Ralph S. Baric對冠狀病毒的RBD關鍵氨基酸,對它們與ACE2,特別是與hACE2的作用機制,就已經研究得極為深入、透徹了;
早在2007,Ralph S. Baric就已經成功實施了關鍵氨基酸的人為替換,成功實施了冠狀病毒的人類組織適應性培養,成功改造、培養出了能感染人體細胞的實驗室病毒。
本系列三篇文章的結論
1)有人參照WIV1(rs3367)病毒的關鍵氨基酸,通過氨基酸復用及等價、等效替換,為新冠病毒設計了一套與WIV1的關鍵氨基酸高度等價、等效的關鍵氨基酸。
2)新冠擁有與WIV1高度等價、等效的關鍵氨基酸,導致了以下“巧合”:
a)新冠、WIV1的跨物種感染能力都極為強大,難分伯仲;
b)新冠、WIV1的宿主範圍都極為廣泛,而且,它們的宿主範圍高度雷同(它們可感染的物種高度重合);
c)新冠、WIV1的刺突蛋白都極為適宜結合hACE2,與hACE2的結合親和力都極為強大。
3)新冠病毒關鍵氨基酸的設計者,即新冠病毒的設計者,極有可能就是Ralph S. Baric,或者Ralph S. Baric團隊的某個、某些資深成員。
























