本文是
谁设计、制造了新冠病毒(三 上)
续文的一部分,为避免续文篇幅过长,现将这部分内容摘出来另成一文。
冠状病毒刺突蛋白RBD中有五个重要位点,这五个位点上的氨基酸残基决定刺突蛋白的ACE2结合能力,进而决定冠状病毒通过结合ACE2受体进入、感染细胞的能力,它们被称为(ACE2相关的)RBD关键氨基酸残基,简称RBD关键氨基酸或RBD关键残基。RBD即受体结合域(Receptor Binding Domain),它是冠状病毒刺突蛋白(spike protein,简称S蛋白)S1亚基的一部分。
“谁设计、制造了新冠病毒(三 上)”指出:新冠病毒的RBD关键氨基酸借鉴自蝙蝠冠状病毒rs3367(WIV1),新冠、rs3367(WIV1)的5个RBD关键氨基酸三个相同,另有一对理化属性高度相似。
下面是Ralph S. Baric(团队)研究RBD关键氨基酸的四个论文实例,前两篇论文的介绍较长,读者可根据个人兴趣择要阅读、跳跃阅读。
论文B-1
2007年12月19日,Ralph
S. Baric领导的团队在Journal of Virology(病毒学杂志)在线发表了如下论文:Mechanisms of
Zoonotic Severe Acute Respiratory Syndrome Coronavirus Host Range
Expansion in Human Airway Epithelium(人畜共患SARS冠状病毒在人气道上皮中扩大宿主范围的机制)
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2258931/
https://journals.asm.org/doi/10.1128/JVI.02041-07
这篇论文需要介绍的内容颇多,展开之前,先概括一下论文的两大要点:
1、果子狸冠状病毒SZ16不能感染人类和灵长类动物;人为替换SZ16的第三关键氨基酸,将所得中间病毒放到人气道上皮中培养,促其进一步发生适应性突变,得到了两种可跨物种感染、传播的冠状病毒,这两种病毒都能感染人气道上皮细胞和灵长类动的Vero E6细胞。
2、深入研究了冠状病毒RBD与hACE2的作用机制,分析、解读了RBD关键氨基酸替换、变异,如何影响、改变ACE2结合能力的内在机理。
下面看论文具体细节。
论文病毒改造的原材料之一是果子狸(civet,有时译作麝香猫)类SARS冠状病毒SZ16(SARS-CoV SZ16),它分离于SARS疫情期间,分离自中国广东地区动物市场中的果子狸。
SARS病毒既能感染人类,也能感染果子狸,但SZ16只能感染果子狸,不能感染人类。为解读二者感染能力不同的内在机制,论文列出了SZ16、两种SARS流行病毒株SARS Urbani、SARS GD03三者刺突蛋白的所有氨基酸差异,见下图:
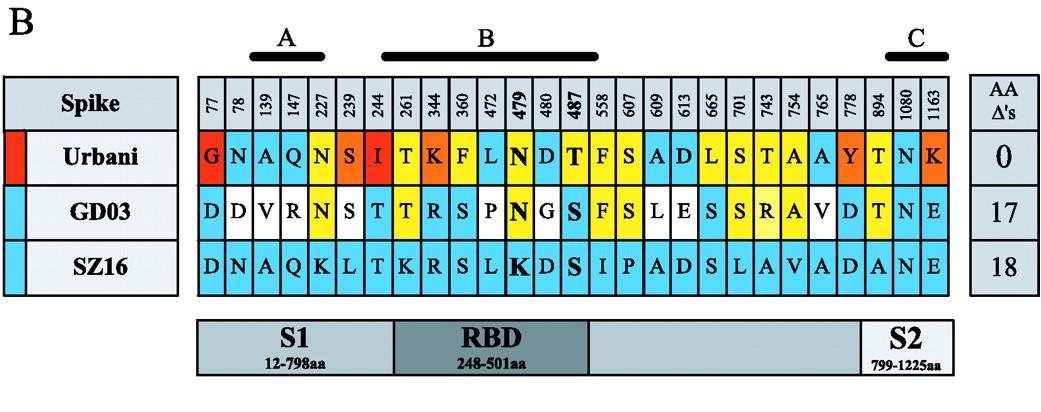
Urbani、GD03、SZ16的刺突蛋白氨基酸差异
图中蓝色标示动物来源的氨基酸(与SZ16中氨基酸一致的氨基酸),黄色标示SARS疫情早期出现的氨基酸,橙棕色标示疫情中期出现的氨基酸,红棕色标示疫情晚期出现的氨基酸。
SZ16与SARS Urbani有18个刺突蛋白氨基酸差异,其中16个不同氨基酸位于S1亚基,5个位于RBD,二者有两个不同RBD关键氨基酸(见上图中加粗加黑的第479、487氨基酸)。
刺突蛋白包括S1亚基、S2亚基两部分,S1亚基是受体结合亚基,S2亚基是膜融合亚基,RBD即受体结合域是S1亚基的一部分。
SARS Urbani是分离于SARS疫情晚期,分离自美国的一个SARS病毒株;
SARS GD03是更晚出现的SARS病毒株,它分离于2004年1月疫情再次零星出现时,分离自一位散发广东患者。
2002~2003年的SARS疫情发生于2002年11月16日-2003年9月2日,高峰期为2002年11月16日-2003年7月16日。
如前所述,SZ16与SARS Urbani有两个RBD关键氨基酸不同。论文将证明,以SZ16的刺突蛋白为原材料,通过改变其RBD关键氨基酸,可以改造、培育出有人体细胞感染能力的新病毒。具体的实施过程如下。
1、用SZ16的刺突蛋白和SARS Urbani的骨架(刺突蛋白外部分)合成了嵌合病毒icSZ16-S。
合成嵌合病毒icSZ16-S的更多细节是这样的:用SZ16刺突蛋白部分的基因序列,和SARS Urbani骨架部分的基因序列,拼出一个嵌合基因序列,而后基于嵌合基因序列,使用反向遗传平台合成嵌合病毒的cDNA、RNA,将所得RNA电穿孔释放到培养细胞中,利用RNA的复制能力,最终在培养细胞中收获嵌合病毒的活性毒株。
基于基因序列合成病毒cDNA、RNA,收获病毒毒株,这是反向遗传平台的基本功能。
嵌合病毒icSZ16-S的刺突蛋白来自SZ16,骨架来自SARS Urbani。实验证明,和SZ16一样,icSZ16-S既不能感染Vero E6细胞(非洲绿猴肾细胞系细胞),也不能感染HAE细胞(人气道上皮细胞)。
注:
ic:infectious cDNA,ic前缀代表病毒是反向遗传平台合成的有生物活性的实验室冠状病毒。SZ16-S表示嵌合病毒icSZ16-S的S蛋白来自SZ16,S蛋白即Spike蛋白,也就是刺突蛋白。
HAE细胞:human airway epithelium cells,人气道上皮细胞,包括纤毛上皮细胞、非纤毛上皮细胞以及杯状细胞。
2、接下来要做RBD关键氨基酸的替换了。研究者将icSZ16-S刺突蛋白的第479氨基酸--赖氨酸K替换为天冬酰胺N(亦称为天门冬酰胺),人为制造了一个氨基酸突变K479N。赖氨酸K是SZ16的第479氨基酸,天冬酰胺N是SARS的第479氨基酸,第479氨基酸是SARS及SZ16五个RBD关键氨基酸中的第三个。因此,K479N就是将icSZ16-S的第三关键氨基酸替换为SARS的第三关键氨基酸。
SZ16的第479氨基酸是赖氨酸K,SARS Urbani、GD03的第479氨基酸是天冬酰胺N。
3、对icSZ16-S施以K479N替换得到了另一个改造病毒:icSZ16-S K479N(要使用反向遗传平台,基于替换后的基因序列将其合成)。简明地说,把icSZ16-S 的479氨基酸由赖氨酸K替换为天冬酰胺N,就得到了icSZ16-S K479N。实验证明,相比icSZ16-S,icSZ16-S K479N对Vero E6细胞和HAE细胞的感染能力有所“进步”,它能感染Vero E6细胞和HAE细胞(人气道上皮细胞),不过只能在两种细胞中低水平地复制,衰弱地传代。
4、为提升icSZ16-S K479N对HAE细胞的适应能力,研究者将其接种到人气道上皮组织中,按一定的策略、方式进行培养,并在其反复传代的过程中加以筛选优化。
培养、传代、筛选的第8天,得到了一个新病毒icSZ16-S K479N D8。相比icSZ16-S K479N,D8发生了一个适应性变异Y442F,即,其刺突蛋白第442氨基酸由酪氨酸Y变异为苯丙氨酸F;
培养、传代、筛选的第22天,又得到了一个新病毒icSZ16-S K479N D22。D22在Y442F变异的基础上,又发生了另一处适应性变异L472F,即,其刺突蛋白第472氨基酸由亮氨酸L变异为苯丙氨酸F。
刺突蛋白的第442氨基酸是SARS和SZ16的第一个RBD关键氨基酸,第472氨基酸是它们的第二个RBD关键氨基酸。也就是说,人为替换icSZ16-S的第三RBD关键氨基酸后得到的中间病毒icSZ16-S K479N,在实验室环境下的人气道上皮组织中,又发生了另外两个RBD关键氨基酸,第一、第二RBD关键氨基酸的适应性突变。
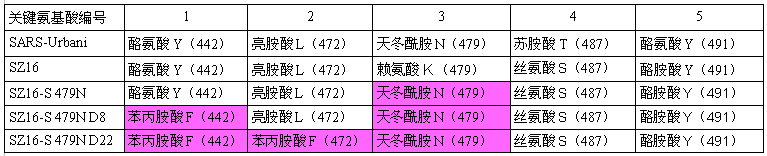
相关病毒RBD关键氨基酸对照表,表中展示了K479N替换、Y442F变异、L472F变异所涉及的RBD关键氨基酸
有一个小小的巧合,新冠病毒、WIV1病毒、icSZ16-S K479N D22病毒的第二个RBD关键氨基酸都是苯丙氨酸F。
新冠、WIV1、SARS、D22四病毒RBD关键氨基酸对照表
论文以四组体外细胞实验证明:
1、icSZ16-S K479N D22和icSZ16-S K479N D8都能有效感染HAE细胞、Vero E6细胞、DBT-hACE2细胞,并在细胞中有效复制。
2、D8在三种细胞中的复制能力或多或少弱于D22,D22在三种细胞中的复制能力又或多或少弱于icSARS(使用反向遗传平台合成的SARS流行毒株Urbani的克隆毒株)。
3、icSZ16-S K479N在HAE细胞和DBT-hACE2中的复制能力低下,复制滴度与D22、D8相差好几个数量级(即D22、D8在两种细胞中的复制能力大大强于K479N)。
4、icSARS、icSZ16-S K479N、icSZ16-S K479N D8、icSZ16-S K479N D22都不能感染未转基因的普通DBT细胞。
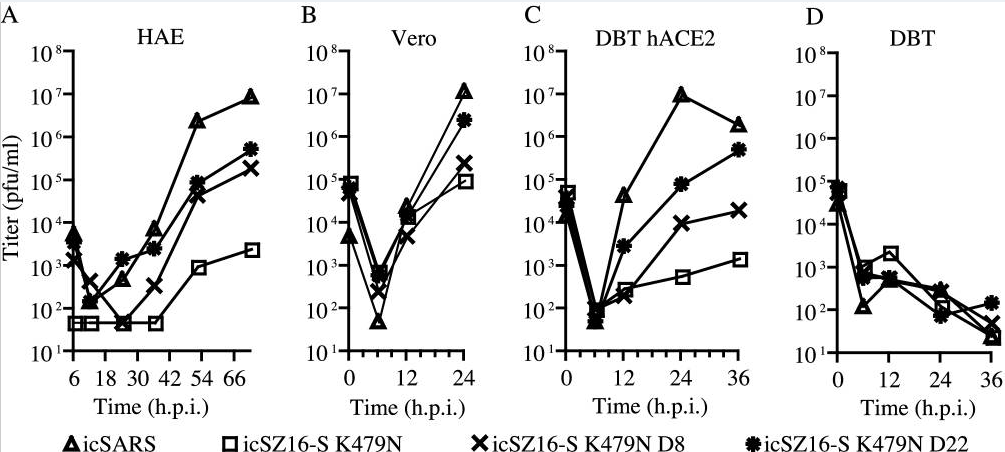
icSARS、K479N、D8、D22对HAE、Vero E6、DBT-hACE2、普通DBT细胞的感染实验数据图。
横坐标:感染后时间,单位为小时;纵坐标:细胞培养物中的病毒滴度,单位为PFU/ml。
注:
PFU,Plaque-forming unit,单位体积的病毒形成的空斑数,是代表有活性的病毒粒子浓度的单位。空斑(Plaque)是病毒诱导宿主细胞裂解形成的点状物,可通过显微镜观察。
DBT-hACE2是Ralph S. Baric团队设计、制造的一种表达hACE2的转基因小鼠DBT细胞,其细胞的跨膜糖蛋白--ACE2受体被转基因为hACE2。
DBT:Delayed Brain Tumor,延迟脑肿瘤;小鼠DBT细胞,即小鼠延迟脑肿瘤细胞,或称为小鼠星形细胞瘤迟发性脑瘤细胞。
论文还以小鼠体内实验证明:D8和D22还能感染BALB/c小鼠(白变种实验室小鼠)的肺细胞,并在小鼠肺中良好生长、复制,不过,它们都不会使BALB/c小鼠产生临床疾病症状。
可见:
1、D8和D22是具有跨物种感染能力的病毒,它们能有效感染人气道上皮细胞,灵长类动物的Vero E6细胞,小鼠的肺细胞。
2、通过替换、改造、改变RBD关键氨基酸,可以改变刺突蛋白的ACE2结合能力,改变冠状病毒的细胞感染能力,扩大其宿主范围,可以将不能感染人类,不能跨物种感染的冠状病毒改造为可感染人类,能够跨物种感染的冠状病毒。早在2007年,Ralph S. Baric就已成功尝试了相关技术。
论文使用蛋白质结构设计、预测工具Rosetta Design生成了SZ16、icSZ16-S K479N、icSZ16-S K479N D8和icSZ16-S K479N D22等病毒的RBD结构模型,并将这些结构模型叠加到SARS Urbani RBD与hACE2相互作用的晶体结构模型上,得到了上述四种病毒与hACE2相互作用的晶体结构模型。
论文借助晶体结构模型研究了病毒RBD与hACE2的作用、结合机制,通过对照这些病毒刺突蛋白第442、472、479、487四个位点的氨基酸差异及与hACE2的作用差异,解读了氨基酸差异影响hACE2结合能力的机理,解释了这些病毒的不同细胞感染能力。上述四个氨基酸是相关各病毒的第一、第二、第三、第四RBD关键氨基酸;论文未对照这些病毒的第五RBD关键氨基酸,即刺突蛋白的第491氨基酸,因为该位点的氨基酸极为保守,论文中各病毒的第五RBD关键氨基酸全都相同,都是酪氨酸Y。具体解读细节见下(供有兴趣者参考,普通读者可跳过):
a)SZ16与hACE2作用的晶体模型图B显示,SZ16的第479氨基酸残基—Lys479(赖氨酸K),似乎与邻近的两个hACE2氨基酸残基Lys31、His34(His代表组氨酸)存在碰撞(静电排斥),导致SZ16的刺突蛋白不能与hACE2良好结合。图B中Lys31和His34周围的点密集团块为深褐色,模型中的深褐色团块表示相应的hACE2残基与附近的RBD残基存在冲突(静电排斥或碰撞)。
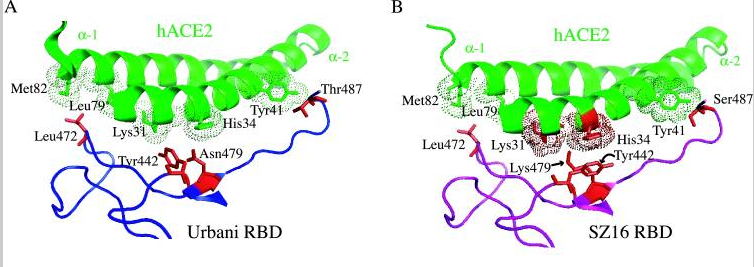
b)icSZ16-S K479N与hACE2作用的晶体模型图C显示,引入点突变K479N后,即将Lys479 (赖氨酸K)替换为Asn479(天冬酰胺N)后,479位点与hACE2残基的排斥力被消除,使得icSZ16-S K479N的刺突蛋白能够与hACE2结合。图C中Lys31和His34两个hACE2残基周围的团块为浅绿色,模型中的浅绿色团块表示相应的hACE2残基与周围的RBD残基相互吻合。
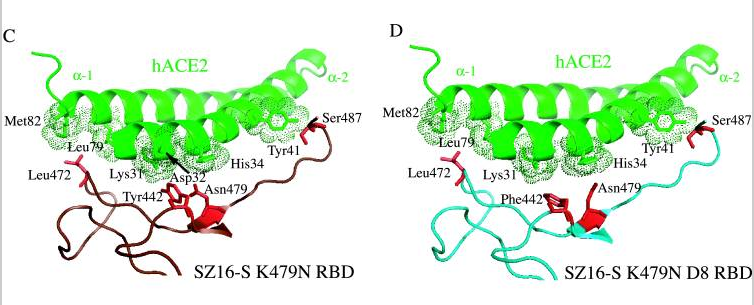
c)前面的晶体模型图A显示,SARS Urbani 的两个RBD关键氨基酸残基Tyr442、Asn479各自与不同的hACE2残基发生作用(Tyr442与Lys31相互作用,Asn479与His34相互作用),二对作用互不干扰,互不竞争,因而Urbani刺突蛋白与hACE2的结合亲和力较强;尽管icSZ16 K479N 的442、479残基与Urbani相同,但软件预测(见上面的晶体模型图C),它的Tyr442、Asn479会同时与hACE2残基Asp32发生作用,同时,Tyr442残基还和两个hACE2残基Asp32、Lys31同时发生作用。这种RBD残基与ACE2残基作用时的交叉竞争削弱了RBD与hACE2的结合亲和力,因而,icSZ16 K479N的刺突蛋白虽然能与hACE2结合,但结合能力较弱。
d)icSZ16-S K479N(以下简称K479N)、SARS Urbani二者唯一不同的RBD关键氨基酸是第487氨基酸(SZ16与Urbani有两个不同RBD关键氨基酸479、487),即第四个RBD关键氨基酸(参见前面的“相关病毒关键氨基酸对照表”)。K479N 的第487氨基酸是Ser487(丝胺酸-S),它只与一个hACE2残基Tyr41结合,见晶体模型图C;SARS Urbani 第487氨基酸是Thr487(苏胺酸-T),它独自结合了三个hACE残基,而且没有其它RBD残基交叉竞争与这三个hACE残基的结合,见前面的晶体模型图A(但我在图A中未看到与Thr487作用的三个hACE2残基???)。因而Urbani的Thr487 残基与hACE2间的氢键强于K479N 的Ser487 残基与hACE2间的氢键,这是Urbani的刺突蛋白与hACE2的结合亲和力强于K479N的刺突蛋白与hACE2的结合亲和力的另一个原因。
e)如晶体模型图D所示,Rosetta Design预测icSZ16-S K479N D8的Y442F突变消除了icSZ16-S K479N的442、479残基对hACE2残基的交叉竞争,D8的Phe442(苯丙氨酸F442)、Asn479各自与不同的hACE2残基作用(Phe442结合Lys31,Asn479结合His34);与此同时,它的Ser487残基与hACE2的Tyr41残基间的相互作用和氢键网络保持不变。因此,D8的hACE2结合能力大大提升,它在HAE细胞和DBT-hACE2细胞中的生长(感染、复制)比K479N茁壮得多。
f)仍如晶体模型图D所示,Rosetta Design还预测icSZ16-S K479N D8的Leu472(亮氨酸L472)残基无交叉竞争地同时结合了两个hACE2残基Leu79、Met82(Met代表甲硫胺酸M);icSZ16-S K479N D22是D8发生L472F突变(亮氨酸L突变为苯丙氨酸F)得到的,如下面的晶体模型图E所示,Rosetta Design预测其Phe472(苯丙氨酸F472)残基将无交叉竞争地同时结合三个hACE2残基Leu79、Met82、Tyr83。Rosetta Design还预测Phe472与这三个hACE2残基间存在强烈的疏水相互作用,进一步加强了彼此的结合。因此,L472F突变增强了D22 RBD与hACE2的结合亲和力,提升了D22对HAE细胞、DBT-hACE2细胞的感染、复制能力(相比D8)。
可见,早在2007年,Ralph S. Baric就掌握了通过RBD关键氨基酸替换、变异,改变ACE2结合能力、细胞感染能力,扩大宿主范围的技术,并深入研究了这一技术的内在机制。
在论文Disscussion部分,研究者意犹未尽地写道:
显然,可能存在多种遗传途径来允许人畜共患病SARS冠状病毒宿主范围的扩大,确定其他接触界面点突变是否能增强人畜共患病病毒对HAE细胞(人气道上皮细胞)的感染将是一件有趣的事情。
所谓的“接触界面点”,是指RBD中与ACE2发生直接接触、作用的14个氨基酸残基,RBD关键氨基酸就是这14个RBD接触残基中对ACE2结合能力起决定作用的五个氨基酸残基。论文人为制造了第三RBD关键氨基酸的K479N突变,上面一段话的言下之意,是可以尝试以制造其它RBD关键氨基酸的人为突变作为病毒改造、培育的起点。
论文B-2
2008年9月1日(Published
online 2008 Jun 25),Ralph S. Baric领导的团队在Journal of
Virology发表了如下论文:Pathways of Cross-Species Transmission of Synthetically
Reconstructed Zoonotic Severe Acute Respiratory Syndrome
Coronavirus(人畜共患严重急性呼吸综合症冠状病毒的跨物种传播途径)
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2519660/
https://journals.asm.org/doi/10.1128/JVI.00818-08
论文B-2是论文B-1研究的后续。
论文研究了7种病毒对3种细胞的感染、复制能力,探讨、解读了病毒的细胞感染、复制能力差异与RBD关键氨基酸-ACE2作用差异之间的内在关联。7种病毒是:
病毒1:论文B-1中的嵌合病毒icSZ16-S,它的刺突蛋白来自果子狸冠状病毒SZ16,骨架来自SARS Urbani;
病毒2:icSZ16-S K479N,它是将icSZ16-S的479氨基酸(赖氨酸K)替换为SARS 的第479氨基酸(天冬酰胺N)得到的病毒;
病毒3:icSZ16-S K479N D22,它是在人气道上皮组织中培养、传代、筛选22天后得到的icSZ16-S K479N的实验室变异体;
病毒4-6:三个SARS病毒株的克隆:icSARS、icCUHK-W1、icGZ02,
icSARS是SARS Urbani的克隆,SARS Urbani是SARS疫情晚期分离自美国的SARS病毒株,
GZ02是SARS疫情早期分离自广州的SARS病毒株,
CUHK-W1是SARS疫情中期分离自香港的SARS病毒株。
病毒7:icGD03-S,它应该是由SARS GD03的刺突蛋白与SARS Urbani的骨架嵌合成的SARS不同毒株的混嵌体。
GD03是更晚出现的SARS病毒株,它是2004年1月疫情再次零星出现时从一位散发广东患者体内分离得到的。
(2002~2003年的SARS疫情发生于2002年11月16日-2003年9月2日,高峰期为2002年11月16日-2003年7月16日)
论文详细考察了7种病毒刺突蛋白(spike)的氨基酸差异,以及刺突蛋白、ACE2的相互作用残基。
图D:相关病毒的spike蛋白氨基酸差异;图E:ACE2、SARS-Urbani的所有接触残基
图D列出了7种病毒间的所有氨基酸差异,并标出了其它6种病毒相对于SARS Urbani的不同刺突蛋白氨基酸个数。如icSZ16-S K479N D22(即图中的SZ16-S K479N D22)有19刺突蛋白氨基酸与SARS Urbani不同(不相同的氨基酸残基背景为灰色)。
图E列出了与SARS Urbani的spike发生接触或作用的所有cACE2(civet ACE2)、hACE2/primate-ACE2(灵长类动物ACE2)氨基酸残基,及与各个ACE2残基发生作用的SARS Urbani的接触残基(氨基酸残基)。
论文B-1研究、实验了相关病毒对HAE细胞(人气道上皮细胞)、Vero E6细胞、普通小鼠DBT细胞、转基因小鼠DBT-hACE2细胞的感染、复制能力。当前论文(B-2)引入了另一种转基因小鼠DBT细胞:DBT-cACE2,它的mACE2(mice ACE2)转基因为cACE2(civet ACE2,果子狸或麝香猫的ACE2)。
论文设计、实施了三组体外感染实验,7种病毒对3种细胞DBT-cACE2 、DBT-hACE2、Vero E6的感染、复制实验数据见下图:
7种病毒对3种细胞的感染复制实验数据图
横坐标是感染后时段,单位为小时;纵坐标是病毒的复制滴度,单位为PFU/ml。
实验结果表明:
1、icSZ16-S K479N、icSZ16-S K479N D22和icCUHK-W1要么不能在DBT-cACE2细胞中有效复制,要么只能低水平复制(滴度小于10^3);而其它4种病毒,包括icSZ16-S在内都能在DBT-cACE2细胞中有效复制(滴度大于10^4),见实验数据图中的坐标图A;
2、icSZ16-S不能在DBT-hACE2细胞中有效复制(滴度约为10^2),而其它6种病毒都能在DBT-hACE2细胞中不同程度地复制(K479N复制能力相对最差,滴度略低于10^4),见坐标图B;
3、icSZ16-S不能在Vero E6细胞中有效复制(滴度仅略高于10^2),而其它6种病毒都能在Vero E6细胞中不同程度地复制(滴度大于10^5),GZ02、CUHK-W1、icSARS、icSZ16-S K479N D22这四种病毒在Vero E6细胞中的复制都极为强烈(滴度大于10^7),见坐标图C;
4、icSZ16-S K479N D22对Vero E6、DBT-hACE2的感染、复制能力强于icSZ16-S K479N ;K479N 替换使icSZ16-S K479N、icSZ16-S K479N D22获得了对Vero E6、DBT-hACE2的感染、复制能力,但同时使它们丧失了icSZ16-S所具有的对DBT-cACE2细胞的感染、复制能力。
论文还指出:上述7种病毒都不能感染未转基因的普通小鼠DBT细胞。
icSZ16-S、icSZ16-S K479N、icSZ16-S K479N D22三者不同物种ACE2结合能力,不同物种细胞感染、复制能力的显著差异是它们RBD关键氨基酸的差异造成的。

相关病毒RBD关键氨基酸对照表
为进一步研究RBD与cACE2、hACE2作用的内在机制,解读RBD关键氨基酸差异对物种ACE2结合能力影响的内在机理,论文使用RosettaDesign和Modeler两种软件构建了4种病毒(SARS Urbani、SZ16、icSZ16-K479N、SZ16-K479N D22)的刺突蛋白与cACE2、hACE2的作用模型(二维、三维结构模型)。以下为论文所作解读(供有兴趣者参考,普通读者可略过):
1、cACE2、hACE2有两个不同的RBD接触残基(与RBD接触的ACE2氨基酸残基)(cACE2的E30、Y34在hACE2中为D30、H34),模型预测,E30和Y34的附加甲基可能会使cACE2结合界面产生明显的突起(我在模型图B下部的三维图中目测看不出cACE2的明显突起)。模型图A显示,Urbani的RBD能良好适应hACE2的结合界面(三维模型图用浅黄绿色表示与RBD良好吻合的ACE2残基);模型图B显示,Urbani也能容纳cACE2的表面突起(三维模型图用浅棕色表示与RBD存在冲突,但仍能结合的ACE2残基)。因此,SARS Urbani对人类和civet(果子狸或麝香猫)有双重宿主适应能力。
SARS Urbani与hACE2、cACE2,SZ16与cACE2作用模型
2、如模型图C所示,SZ16 的RBD也能容纳E30、Y34形成的表面突起,因而SZ16 的RBD也可与cACE2结合。
3、如模型图D所示,icSZ16 K479N中的K479N突变(人为替换)引入的479N(天冬酰胺)重塑了SZ16刺突蛋白的结合界面,促进了刺突蛋白与hACE2的结合(论文为什么不提供、对照SZ16 RBD与hACE2的作用模型?)。
icSZ16 K479N与hACE2、cACE2,icSZ16 K479N D22与hACE2的作用模型
4、如模型图E所示,SZ16 K479N RBD的结合界面因K479N替换而被重塑,它的氨基酸残基(V404、Y440)与cACE2的氨基酸残基(E30、Y34)产生了冲突,无法容纳cACE2的延长突出部分(三维图中的深褐色部分代表RBD残基与ACE2残基间存在难以克服的冲突,难以产生结合的区域)。从而,K479N突变(人为替换)使SZ16 K479N 的RBD获得了结合hACE2的能力,但同时失去了结合cACE2的能力。
5、如模型图F所示,Y442F突变和L472F突变在K479N突变基础上进一步重塑了SZ16 K479N D22的RBD结合界面,大大提高了RBD与hACE2的吻合度和结合效率;
6、如模型图G所示,和SZ16 K479N一样,SZ16 K479N D22的RBD也不能容纳cACE2的延长突出部分,二者无法结合。
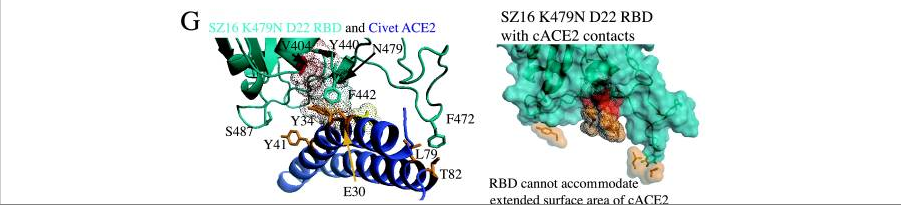
icSZ16 K479N D22与cACE2的作用模型
由论文B-1、B-2可知:
1、早在2007年,Ralph S. Baric就通过人为替换RBD关键氨基酸,结合实验室环境下人体组织适应性培养,改造、培育出了能感染人体细胞,并具有跨物种感染、传播能力的冠状病毒。
2、Ralph S. Baric对冠状病毒RBD(特别是RBD关键氨基酸)与不同物种ACE2的作用机制,早在2007、2008年就已经研究得极为深入了。
论文B-3
2015年11月9日,Ralph
S. Baric领导的团队在《自然医学》(Nature Medicine)杂志发表了著名的嵌合病毒论文:A SARS-like cluster
of circulating bat coronaviruses shows potential for human
emergence(一个类似SARS的蝙蝠冠状病毒群显示了产生人类流行疫情的潜力)
https://www.nature.com/articles/nm.3985
论文同时研究了WIV1病毒和RBD关键氨基酸,它也将作为Ralph S. Baric研究rs3367(WIV1)的示例(论文A-1)被介绍。下面从RBD关键氨基酸的角度简单介绍一下该论文。
WIV1在论文中是病毒主角SHC014的对照病毒。论文比较了WIV1、SHC014与SARS的RBD关键氨基酸差异。论文(援引其它论文的实验结果)指出:WIV1虽然有三个RBD关键氨基酸与SARS不同(见下图,二者第1、第2、第4 RBD关键氨基酸不同),但WIV1仍象SARS那样,其刺突蛋白也能有效结合hACE2;论文接着指出,尽管SHC014的RBD关键氨基酸与SARS差异更大,二者的RBD关键氨基酸无一相同,但不能就此断言SHC014的刺突蛋白不能结合hACE2。
SARS、WIV1、SHC014 RBD关键氨基酸对照表
为确定SHC014的hACE2结合能力、人体细胞感染能力,论文使用反向遗传平台,基于SHC014刺突蛋白(的基因序列)与SARS-CoV-MA15骨架(的基因序列)合成了嵌合病毒SHC014-MA15。实验证明,SHC014-MA15能有效感染人气道上皮组织,并在组织细胞内大量复制。这表明,SHC014的刺突蛋白也能有效结合hACE2,和WIV1一样,SHC014也是具有人体细胞进入(感染)能力的特殊蝙蝠冠状病毒。
论文B-4
2016年3月14日,Ralph S. Baric领导的团队在PNAS(美国国家科学院院刊)发表了如下论文:SARS-like WIV1-CoV poised for human emergence(类SARS冠状病毒WIV1-CoV对人类有潜在威胁)
https://www.pnas.org/content/113/11/3048
论文B-4也同时研究了WIV1病毒和RBD关键氨基酸,它还将作为Ralph S. Baric研究rs3367(WIV1)的示例(论文A-2)被介绍。
论文比较了SARS-WT、SARS-MA15、WIV1三病毒与hACE2直接作用(接触)的14个RBD氨基酸残基,包括五个RBD关键氨基酸残基,见下图A;论文构建、研究了SARS、WIV1的刺突蛋白与hACE2的作用模型,对照了二者第一、第二、第四RBD关键氨基酸残基(对应SARS刺突蛋白的第442、472、487氨基酸)与hACE2的作用细节,见下图B。
图A:WIV1、SARS 14个ACE2直接作用残基对照表;图B:WIV1、SARS 与ACE2的作用模型对照图
最后是对图A、图B的一些补充说明:
1、图A中的氨基酸序号基于SARS,WIV1中的对应氨基酸序号应+1。
2、SARS-WT,即SARS-CoV wild type,指SARS病毒的野生流行毒株(它应该具体对应SARS Urbani)。
3、SARS-MA15,即SARS-CoV MA15,Ralph S. Baric等人实验室培育的SARS-CoV小鼠适应性变异体。SARS-CoV能感染小鼠细胞,但不会使小鼠产生临床疾病症状,SARS-CoV MA15则可使小鼠严重致病并(100%)致死。
4、图A中,SARS-MA15与SARS-WT不同的氨基酸用深红背景色标示,WIV1-CoV与SARS-WT不同的氨基酸用深蓝背景色标示,紫红色长条框标出了三病毒的5个RBD关键氨基酸残基。
5、图B左图是SARS刺突蛋白S1亚基(也叫受体结合亚基,S1亚基前端为RBD)与hACE2的作用模型,图B右图是WIV1刺突蛋白S1亚基与hACE2的作用模型。
左右两图右侧的双螺旋结构是hACE2,其上的橙黄色部分是hACE2的RBD接触残基;
左右两图左侧是两病毒刺突蛋白的S1亚基,SARS的S1亚基以灰黑色表示,WIV1的S1亚基以蓝色表示。两个红色含环图案对应两病毒相同的RBD关键氨基酸残基(479、491),紫色圆圈标出了二者不同的三个RBD关键氨基酸残基(442、472、487)与hACE2残基的作用情况。




















