| 鐵證如山:石正麗造了Covid-19!(中文翻譯) |
| 送交者: 群山 2023年05月31日04:38:07 於 [五 味 齋] 發送悄悄話 |
|
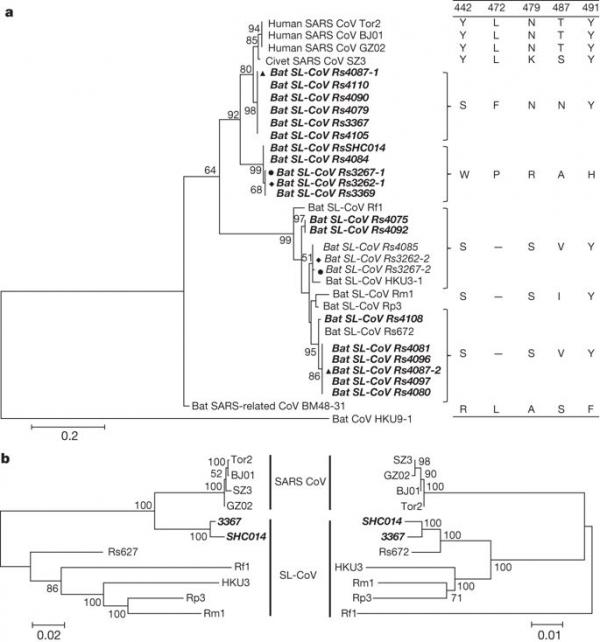
Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor • Xing-Yi Ge, • Jia-Lu Li, • Xing-Lou Yang, • Aleksei A. Chmura, • Guangjian Zhu, • Jonathan H. Epstein, • Jonna K. Mazet, • Ben Hu, • Wei Zhang, • Cheng Peng, • Yu-Ji Zhang, • Chu-Ming Luo, • Bing Tan, • Ning Wang, • Yan Zhu, • Gary Crameri, • Shu-Yi Zhang, • Lin-Fa Wang, • Peter Daszak & • Zheng-Li Shi(石正麗) Nature 第 503 卷,第 535–538 頁(2013)引用這篇文章 • 59k 次訪問 • 299 次引用 • 1125 Altmetric • 指標詳情 摘要 2002-3 年由嚴重急性呼吸系統綜合症冠狀病毒 (SARS-CoV) 引起的大流行是近代歷史上最重大的公共衛生事件之一。 中東呼吸綜合徵冠狀病毒 2 的持續爆發表明,這組病毒仍然是一個主要威脅,而且它們的分布範圍比以前認識到的要廣。 儘管蝙蝠被認為是這兩種病毒的天然宿主3、4、5,但從蝙蝠中分離出 SARS-CoV 的祖病毒的嘗試並未成功。 現在已經從中國、歐洲和非洲的蝙蝠中報道了多種 SARS 樣冠狀病毒 (SL-CoV)5,6,7,8,但由於它們與該病毒的系統發育差異以及 他們的刺突蛋白無法使用 SARS-CoV 細胞受體分子,即人血管緊張素轉換酶 II (ACE2)9,10。 在這裡,我們報告了來自中國雲南中國馬蹄蝠(家族:Rhinolophidae)的兩種新型蝙蝠冠狀病毒的全基因組序列:RsSHC014 和 Rs3367。 這些病毒與 SARS-CoV 的關係比之前發現的任何蝙蝠冠狀病毒都要密切,特別是在刺突蛋白的受體結合域。 最重要的是,我們首次報告了在 Vero E6 細胞中從蝙蝠糞便樣本中分離出活 SL-CoV(蝙蝠 SL-CoV-WIV1)的記錄,它具有典型的冠狀病毒形態,與 Rs3367 的序列同一性為 99.9%,並使用來自人類的 ACE2, 果子狸和中國馬蹄蝙蝠進入細胞。 初步體外試驗表明,WIV1 還具有廣泛的物種嗜性。 我們的研究結果提供了迄今為止最有力的證據,證明中國馬蹄蝠是 SARS-CoV 的天然宿主,中間宿主可能不是某些蝙蝠 SL-CoV 直接感染人類所必需的。 他們還強調了針對新興疾病熱點地區高風險野生動物群體的病原體發現計劃作為大流行防範戰略的重要性。 主要的 2002-3 年的 SARS1 大流行和中東呼吸綜合徵冠狀病毒 (MERS-CoV)2 的持續出現表明,CoV 是一種重大的公共衛生威脅。 SARS-CoV 被證明使用人類 ACE2 分子作為其進入受體,這被認為是其跨物種傳播能力的標誌 11。 位於 SARS-CoV 刺突 (S) 蛋白氨基末端區域(氨基酸 318-510)的受體結合域 (RBD) 直接參與與 ACE2 的結合(參考文獻 12)。 然而,儘管系統發育證據表明 SARS-CoV 是從蝙蝠 SL-CoV 進化而來的,但所有先前發現的 SL-CoV 在其 S 蛋白的 RBD 中都與 SARS-CoV 存在主要序列差異,包括一兩個缺失 6,9。 用 SARS-CoV S 替換一種 SL-CoV S 蛋白的 RBD 賦予了使用人類 ACE2 並在小鼠中有效複製的能力9,13。 然而,迄今為止,還沒有從蝙蝠中分離出 SL-CoV,也沒有發現源自蝙蝠的野生型 SL-CoV 使用 ACE2。 我們在中國雲南省昆明市的一個地點對中華犀牛群落中的 SL-CoV 進行了為期 12 個月的縱向調查(2011 年 4 月至 2012 年 9 月)(擴展數據表 1)。 使用先前公布的方法 5、14 從單個蝙蝠身上收集了總共 117 個肛拭子或糞便樣本。 進行了一步逆轉錄 (RT) 套式 PCR 以擴增 RNA 依賴性 RNA 聚合酶 (RdRP) 基序 A 和 C,它們在甲型冠狀病毒和乙型冠狀病毒中是保守的15。 117 個樣本中有 27 個 (23%) 通過 PCR 被歸類為陽性,隨後通過測序確認。 如前所述 16,通過細胞色素 b 序列分析,確認所有陽性樣品的物種來源為 R. sinicus。 在 10 月收集的樣本中觀察到更高的患病率(2011 年為 30%,2012 年為 48.7%)高於 4 月(2011 年為 7.1%)或 5 月(2012 年為 7.4%)(擴展數據表 1)。 S 蛋白 RBD 序列分析表明存在七種不同的 SL-CoV 菌株(圖 1a 和擴展數據圖 1 和圖 2)。 除了與先前描述的 SL-CoV(Rs672、Rf1 和 HKU3)5、8、17、18 密切匹配的 RBD 序列外,還發現了兩種新菌株(命名為 SL-CoV RsSHC014 和 Rs3367)。 確定了它們的全長基因組序列,發現兩者的大小均為 29,787 個鹼基對(不包括 poly(A) 尾巴)。 這兩個基因組與人類 SARS-CoV(Tor2 株)的整體核苷酸序列同一性為 95%,高於之前在中國(88-92%)5,8,17,18 或歐洲觀察到的蝙蝠 SL-CoV( 76%)6(擴展數據表 2 和擴展數據圖 3 和圖 4)。 在這些新的 SL-CoV 和 SARS-CoV 之間的蛋白質水平上觀察到更高的序列同一性(擴展數據表 3 和 4)。 為了解這兩種新型 SL-CoV 毒株的進化起源,我們使用重組檢測程序 4.0 包 19 使用可用的蝙蝠 SL-CoV 毒株基因組序列(Rf1、Rp3、Rs672、Rm1、HKU3 和 BM48-31)進行了重組分析 以及人類和果子狸的代表性 SARS-CoV 毒株(BJ01、SZ3、Tor2 和 GZ02)。 檢測到叄個具有強 P 值 (<10-20) 的斷點,並得到相似圖和引導掃描分析的支持(擴展數據圖 5a、b)。 斷點位於 Rs3367(和 RsSHC014)基因組的第 20,827、26,553 和 28,685 位核苷酸,並生成覆蓋第 20,827–26,533 位核苷酸(5,727 個核苷酸)的重組片段(包括部分開放閱讀框 (ORF) 1b、全長 S、ORF3、 E 和部分 M 基因)和核苷酸 26,534–28,685(2,133 個核苷酸)(包括部分 ORF M、全長 ORF6、ORF7、ORF8 和部分 N 基因)。 使用主要和次要親本區域的系統發育分析表明,Rs3367 或 RsSHC014 是譜系重組的後代,最終導致 SARS-CoV 和 SL-CoV Rs672(圖 1b)。 圖 1:基於 S RBD 區域和蝙蝠 SL-CoV Rs3367 或 RsSHC014 的兩個親本區域的氨基酸序列的系統發育樹。
a,使用 ClustalW 軟件將 SARS-CoV S 蛋白氨基酸殘基 310-520 與蝙蝠 SL-CoV 的同源區域對齊。 使用泊松模型構建最大似然系統發育樹,其自舉值由 MEGA5 軟件包中的 1,000 次重複確定。 本研究中確定的 RBD 序列以粗體顯示並以樣本編號命名。 與人類 ACE2 分子相互作用的關鍵氨基酸殘基顯示在樹的右側。 SARS-CoV GZ02、BJ01 和 Tor2 分別從 2003 年 SARS 爆發的早期、中期和晚期患者中分離出來。SARS-CoV SZ3 是在 2003 年從中國廣東採集的 Paguma larvata 中鑑定出來的。 SL-CoV Rp3、Rs672 和 HKU3-1 是從中國採集的中華紅斑狼蛛中鑑定出來的(分別為:廣西,2004 年;貴州,2006 年;香港,2005 年)。 Rf1 和 Rm1 分別從 2004 年在中國湖北採集的 R. ferrumequinum 和 R. macrotis 中鑑定出來。Bat SARS 相關冠狀病毒 BM48-31 從 2008 年在保加利亞採集的 R. blasii 中鑑定出來。Bat CoV HKU9-1 是從 2005/2006 年在中國廣東收集的 Rousettus leschenaultii 中鑑定出來的,並用作外群。 在當前研究中確定了所有粗體和斜體序列。 實心叄角形、圓形和菱形表示樣本被兩種不同的 SL-CoV 共同感染。 “-”表示氨基酸缺失。 b,Rs3367 或 RsSHC014 的兩個親本區域的系統發育起源。 最大似然系統發育樹由兩個片段的比對構建,分別覆蓋 Rs3367 基因組的核苷酸 20,827-26,533(5,727 個核苷酸)和 26,534-28,685(2,133 個核苷酸)。 出於顯示目的,這些樹是中點根的。 分類單元根據菌株名稱進行注釋:SARS-CoV、SARS 冠狀病毒; SARS 樣冠狀病毒,蝙蝠 SARS 樣冠狀病毒。 兩種新型 SL-CoV,Rs3367 和 RsSHC014,以粗體和斜體顯示。 這兩種新的 SL-CoV 與之前鑑定的 SL-CoV 之間最顯着的序列差異在於其 S 蛋白的 RBD 區域。 首先,它們與 SARS-CoV 具有更高的氨基酸序列同一性(RsSHC014 和 Rs3367 分別為 85% 和 96%)。 其次,沒有缺失,它們與 SARS-CoV RBD 區域具有完美的序列比對(擴展數據圖 1 和 2)。 結構和誘變研究先前已經確定了 SARS-CoV S 蛋白 RBD 中的五個關鍵殘基(氨基酸 442、472、479、487 和 491),它們在受體結合中具有關鍵作用 20, 21。 儘管發現 RsSHC014 S 蛋白中的所有五個殘基均不同於 SARS-CoV,但 Rs3367 RBD 中的五個殘基中有兩個是保守的(圖 1 和擴展數據圖 1)。 儘管在過去十年中蝙蝠 CoV 序列迅速積累,但還沒有成功分離病毒的報道 6,22,23。 我們嘗試從 SL-CoV PCR 陽性樣本中分離出來。 使用優化的方桉和 Vero E6 細胞,我們獲得了一個在第二次盲傳期間引起細胞病變效應的分離物。 純化的病毒顆粒在電子顯微鏡下顯示出典型的冠狀病毒形態(圖 2)。 使用序列非依賴性擴增方法 14 進行序列分析以避免 PCR 引入的污染表明該分離物與 Rs3367 幾乎相同,具有 99.9% 的核苷酸基因組序列同一性和 S1 區域的 100% 氨基酸序列同一性。 新分離株被命名為 SL-CoV-WIV1。 圖 2:純化病毒體的電子顯微照片。
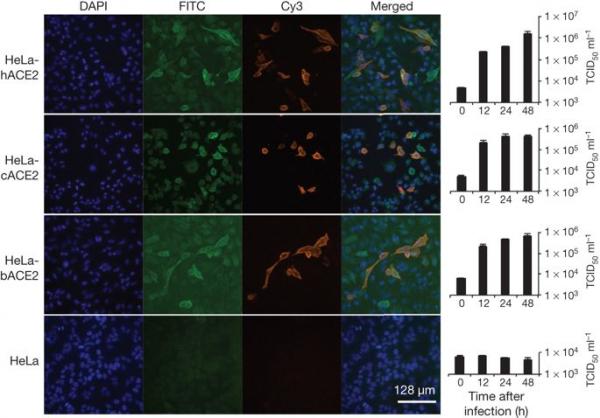
從 10 毫升培養物中收集病毒粒子,固定並通過蔗糖梯度離心法濃縮/純化。 將沉澱的病毒顆粒懸浮在 100 μl PBS 中,用 2% 磷鎢酸 (pH 7.0) 染色,並使用 Tecnai 透射電子顯微鏡 (FEI) 在 200 kV 下直接檢查。 為了確定 WIV1 是否可以使用 ACE2 作為細胞進入受體,我們使用表達或不表達 ACE2 的 HeLa 細胞進行了病毒感染性研究,這些細胞來自人類、果子狸或中國馬蹄蝠。 我們發現 WIV1 能夠使用不同來源的 ACE2 作為進入受體,並在表達 ACE2 的細胞中有效複製(圖 3)。 據我們所知,這是首次鑑定出能夠使用 ACE2 作為進入受體的野生型蝙蝠 SL-CoV。 圖 3:通過免疫熒光測定和實時 PCR 確定的 SL-CoV-WIV1 受體使用分析。
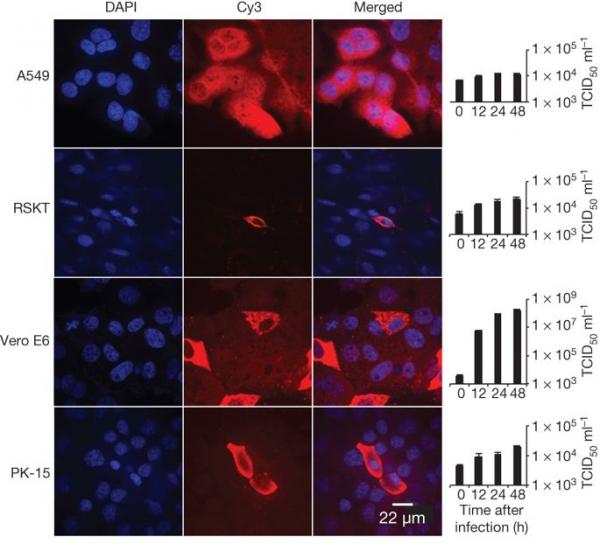
確定表達和不表達 ACE2 的 HeLa 細胞中的病毒感染性。 b、蝙蝠; c、果子狸; 哈,人類。 先用山羊抗人 ACE2 抗體檢測 ACE2 表達,然後用異硫氰酸熒光素 (FITC) 偶聯的驢抗山羊 IgG 檢測。 用針對 SL-CoV Rp3 核衣殼蛋白的兔抗體檢測病毒複製,然後用花青 3 (Cy3) 偶聯的小鼠抗兔 IgG 檢測病毒複製。 細胞核用 DAPI(4',6-二脒基-2-苯基吲哚)染色。 列(從左到右)分別顯示細胞核染色(藍色)、ACE2 表達(綠色)、病毒複製(紅色)、合併的叄重染色圖像和實時 PCR 結果。 (n = 3); 誤差條代表標準偏差。 為了評估其跨物種傳播潛力,我們對一系列物種的細胞系進行了感染性測定。 我們的結果(圖 4 和擴展數據表 5)表明蝙蝠 SL-CoV-WIV1 可以在人肺泡基底上皮細胞 (A549)、豬腎 15 (PK-15) 和 Rhinolophus sinicus 腎 (RSKT) 細胞系中生長,但是 不存在於人子宮頸 (HeLa)、敘利亞金倉鼠腎 (BHK21)、鼠耳鼠腎 (BK)、中華鼠尾草腎 (MCKT)、Rousettus leschenaulti 腎 (RLK) 或 Pteropus alecto 腎 (PaKi) 細胞系中。 實時 RT-PCR 表明 WIV1 在 A549、PK-15 和 RSKT 細胞中的複製效率遠低於在 Vero E6 細胞中的複製效率(圖 4)。 圖 4:通過免疫熒光測定和實時 PCR 確定的 SL-CoV-WIV1 宿主範圍分析。
A549、RSKT、Vero E6 和 PK-15 細胞中的病毒感染。 如圖 3 所述檢測病毒複製。列(從左到右)分別顯示細胞核染色(藍色)、病毒複製(紅色)、合併的雙染色圖像和實時 PCR 結果。 n = 3; 誤差線代表 s.d. 為了評估人 SARS-CoV 血清對 WIV1 的交叉中和活性,我們使用 2003 年收集的 SARS 患者的九份恢復期血清進行了血清中和試驗。結果表明,其中七份能夠完全中和 100 組織培養感染劑量 50 (TCID50) WIV1 以 1:10 至 1:40 的稀釋度,進一步證實了 WIV1 與 SARS-CoV 之間的密切關係。 我們的研究結果對公共衛生具有重要意義。 首先,它們提供了迄今為止最明確的證據表明 SARS-CoV 起源於蝙蝠。 我們之前的工作提供了這方面的系統發育證據 5,但直到現在,由於缺乏蝙蝠 SL-CoV 可以自然感染人類細胞的分離物或證據,人們對這一假設產生了懷疑。 其次,SL-CoV 缺乏使用 ACE2 受體的能力以前被認為是它們直接溢出到人類的主要障礙,這支持了在 SARS 爆發期間果子狸是 SARS-CoV 適應人類傳播的中間宿主的觀點 24 . 然而,SL-CoV-WIV1 使用人類 ACE2 的能力反駁了 SL-CoV-WIV1 這一步的必要性,並表明蝙蝠直接感染人類是某些蝙蝠 SL-CoV 的合理場景。 面對最近發現的具有廣泛地理分布的多樣化且不斷增長的類 SARS 冠狀病毒可能溢出,這對公共衛生控制措施產生了影響。 我們的研究結果表明,蝙蝠冠狀病毒的多樣性大大高於之前報道的多樣性。 在這項研究中,我們能夠證明在 12 個月的時間裡,至少七種不同的 SL-CoV 菌株在單個 R. sinicus 菌落內循環。 該菌落內 SL-CoV 的高度遺傳多樣性反映在不同菌株對 ACE2 的差異使用中的高度表型多樣性。 因此,如果進一步的監測發現能夠使用 ACE2 的蝙蝠 SL-CoV 種類繁多,其中一些與 SARS-CoV 的同源性可能比 SL-CoV-WIV1 更接近,也就不足為奇了。 我們的結果——除了最近在沙特阿拉伯蝙蝠身上發現的 MERS-CoV25,以及在中國、非洲、歐洲和北美 3,26,27 中與 MERS-CoV 密切相關的蝙蝠 CoV——表明蝙蝠冠狀病毒仍然是一個重要的 對全球公共衛生的威脅。 最後,這項研究證明了針對野生動物的病原體發現計劃的公共衛生重要性,這些計劃旨在識別“已知的未知數”——與已知病原體密切相關的以前未知的病毒株。 這些項目專注於特定的高風險野生動物群體和疾病出現的熱點地區,可能是未來預測、準備和預防大流行病出現的全球戰略的重要組成部分 28。 方法總結 如前所述 14,在病毒運輸介質中收集咽喉和糞便拭子或新鮮糞便樣本。 所有 PCR 均使用 One-Step RT-PCR 試劑盒 (Invitrogen) 進行。 如前所述,針對 RdRP 基因高度保守區域的引物用於檢測所有甲型冠狀病毒和乙型冠狀病毒 15。 簡併引物是根據所有可用的 SARS-CoV 和 SL-CoV 基因組序列設計的,用於擴增 S 基因的 RBD 序列或全長基因組序列。 如前所述,簡併引物用於擴增蝙蝠 ACE2 基因 29。 將 PCR 產物凝膠純化並克隆到 pGEM-T Easy Vector (Promega) 中。 至少對四個獨立的克隆進行測序以獲得共有序列。 PCR 陽性糞便樣本(在 200μl 緩衝液中)在 3,000–12,000g 下梯度離心,上清液在 DMEM 中以 1:10 稀釋,然後添加到 Vero E6 細胞中。 在 37°C 孵育 1h 後,取出接種物並用含有 2% FCS 的新鮮 DMEM 代替。 細胞在 37°C 下孵育,並每天檢查細胞病變效應。 來自不同來源的細胞系在 24 孔板的蓋玻片上生長,並以 10 的感染複數接種新型 SL-CoV。感染後 24 小時,使用針對 SL-CoV Rp3 核衣殼蛋白的兔抗體檢測病毒複製 其次是 Cy3 綴合的山羊抗兔 IgG。 在線方法 採樣 如前所述,蝙蝠被困在它們的自然棲息地中 5。 咽喉和糞便拭子樣本收集在病毒轉運培養基 (VTM) 中,該培養基由 Hank 平衡鹽溶液組成,pH 7.4,含有 BSA (1%)、兩性黴素 (15 μg ml−1)、青黴素 G (100U ml−1) 和 鏈黴素 (50μgml−1)。 為了收集新鮮的糞便樣本,每晚約 18:00 將 2.0 x 2.0 米的乾淨塑料布放在已知的蝙蝠棲息地下方。 第二天早上大約 05:30–06:00 從床單上收集相對新鮮的糞便樣本,並將其放入 VTM 中。 樣品被運送到實驗室並儲存在-80℃直至使用。 所有被困在這項研究中的動物在收集樣本後都被釋放回它們的棲息地。 所有採樣過程均由獸醫根據與加州大學戴維斯分校的機構間協議(加州大學戴維斯分校協議編號 16048)獲得武漢病毒研究所動物倫理委員會 (WIVH05210201) 和生態健康聯盟的批准。 RNA提取、PCR和測序 按照製造商的說明,使用病毒 RNA 迷你試劑盒(Qiagen)從 140μl 拭子或糞便樣本中提取 RNA。 RNA 在 60μl 無 RNAse 緩衝液(緩衝液 AVE,Qiagen)中洗脫,然後等分並儲存在 -80℃。 一步 RT-PCR(Invitrogen)用於檢測冠狀病毒序列,如前所述 15。 第一輪 PCR 在含有 12.5μl PCR 2× 反應溷合物緩衝液、10pmol 每種引物、2.5mM MgSO4、20U RNase 抑製劑、1μl SuperScript III/Platinum Taq 酶溷合物和 5μl RNA 的 25μl 反應溷合物中進行第一輪 PCR . RdRP 基因片段的擴增如下進行:50°C 30min,94°C 2min,然後進行 40 個循環,包括 94°C 15s,62°C 15s,68°C for 40s,最後 68°C 延伸 5min。 第二輪 PCR 在含有 2.5μl PCR 反應緩衝液、5pmol 每種引物、50mM MgCl2、0.5mM dNTP、0.1μl Platinum Taq 酶(Invitrogen)和 1μl 第一輪 PCR 產物的 25μl 反應溷合物中進行。 RdRP 基因片段的擴增如下進行:94°C 5min,然後進行 35 個循環,包括 94°C 30s,52°C 30s,72°C 40s,最後延伸 72°C 5min。 為了擴增 RBD 區域,使用基於可用的 SARS-CoV 或蝙蝠 SL-CoV 設計的引物進行一步 RT-PCR(第一輪 PCR 引物;F,正向;R,反向:CoVS931F-5'-VWGADGTTGTKAGRTTYCCT-3 '和CoVS1909R-5'-TAARACAVCCWGCYTGWGT-3';第二個PCR引物:CoVS951F-5'-TGTKAGRTTYCCTAAYATTAC-3'和CoVS1805R-5'-ACATCYTGATANARAACAGC-3')。 第一輪 PCR 在 25 μl 反應溷合物中進行,如上所述,除了使用對 S 基因特異的引物。 S 基因 RBD 區域的擴增如下進行:50°C 30min,94°C 2min,然後 94°C 15s,43°C 15s,68 個循環 35 個循環 °C 90s,最後延伸 68°C 5min。 第二輪 PCR 在含有 2.5μl PCR 反應緩衝液、5pmol 每種引物、50mM MgCl2、0.5mM dNTP、0.1μl Platinum Taq 酶(Invitrogen)和 1μl 第一輪 PCR 產物的 25μl 反應溷合物中進行。 擴增如下進行:94°C 5min,然後 40 個循環,包括 94°C 30s,41°C 30s,72°C 60s,最後延伸 72°C 5min 將 PCR 產物凝膠純化並克隆到 pGEM-T Easy Vector (Promega) 中。 對至少四個獨立的克隆進行測序以獲得每個擴增區域的共有序列。 全長基因組測序 簡併冠狀病毒引物是根據GenBank中所有可用的SARS-CoV和蝙蝠SL-CoV序列設計的,特異性引物是根據本研究前幾輪測序產生的基因組序列設計的(引物序列將根據要求提供)。 所有 PCR 均使用 One-Step RT-PCR 試劑盒 (Invitrogen) 進行。 分別使用 5' 或 3' RACE 試劑盒 (Roche) 確定 5' 和 3' 基因組末端。 將 PCR 產物凝膠純化並直接測序或在克隆到 pGEM-T Easy Vector (Promega) 後測序。 至少對四個獨立的克隆進行測序,以獲得每個擴增區域的共有序列,並且每個區域至少測序兩次。 序列分析和數據庫登錄號 使用 DNAStar 或 Geneious 進行常規序列管理和分析。 使用 ClustalW、BioEdit 或 GeneDoc 進行序列比對和編輯。 基於蛋白質序列的最大似然系統發育樹是使用泊松模型構建的,其引導值由 MEGA5 軟件包中的 1,000 次重複確定。 本研究中獲得的序列已保藏在 GenBank 中,登記號在括號中: WIV1 S (KC881007) 的全長序列; RBD(KC880984-KC881003); ACE2 (KC8810040)。 本研究中使用的 SARS-CoV 序列:人類 SARS-CoV 毒株 Tor2 (AY274119)、BJ01 (AY278488)、GZ02 (AY390556) 和果子狸 SARS-CoV 毒株 SZ3 (AY304486)。 本研究中使用的蝙蝠冠狀病毒序列:Rs672 (FJ588686)、Rp3 (DQ071615)、Rf1 (DQ412042)、Rm1 (DQ412043)、HKU3-1 (DQ022305)、BM48-31 (NC_014470)、HKU9-1 (NC_009021)、HKU4 (NC_009019)、HKU5 (NC_009020)、HKU8 (DQ249228)、HKU2 (EF203067)、BtCoV512 (NC_009657)、1A (NC_010437)。 本研究中使用的其他冠狀病毒序列:HCoV-229E (AF304460)、HCoV-OC43 (AY391777)、HCoV-NL63 (AY567487)、HKU1 (NC_006577)、EMC (JX869059)、FIPV (NC_002306)、PRCV (DQ811787)、BWCoV (NC_010646)、MHV (AY700211)、IBV (AY851295)。 蝙蝠ACE2基因的擴增、克隆與表達 在 pcDNA3.1 中構建人和果子狸 ACE2 的表達克隆之前已有描述29。 Bat ACE2 是從 R. sinicus(樣品編號 3357)中擴增出來的。 簡而言之,使用 RNeasy Mini Kit (Qiagen) 從蝙蝠直腸組織中提取總 RNA。 通過用隨機六聚體逆轉錄從總 RNA 合成第一鏈互補 DNA。 使用正向引物 bAF2 和反向引物 bAR2 擴增全長蝙蝠 ACE2 片段(參考文獻 29)。 用KpnI和XhoI將ACE2基因克隆到pCDNA3.1中,測序驗證。 將純化的 ACE2 質粒轉染至 HeLa 細胞。 24 小時後,表達人、果子狸或蝙蝠 ACE2 的 HeLa 細胞裂解物通過蛋白質印跡或免疫熒光測定法進行確認。 蛋白質印跡分析 細胞裂解物或含有假病毒的過濾上清液通過 SDS-PAGE 分離,然後轉移到硝酸纖維素膜 (Millipore)。 為了檢測 S 蛋白,將膜與兔抗 Rp3 S 片段(氨基酸 561–666)多抗體(1:200)一起孵育,並通過鹼性磷酸酶 (AP) 偶聯的山羊抗兔 IgG 檢測結合的抗體 (1:1,000)。 為了檢測上清液中的 HIV-1 p24,使用抗 HIV p24 單克隆抗體 (p24 MAb) 作為一抗,稀釋度為 1:1,000,然後與 AP 偶聯山羊抗小鼠 IgG 以相同稀釋度孵育。 為了檢測 HeLa 細胞中 ACE2 的表達,使用抗人 ACE2 胞外域的山羊抗體(1:500)作為一抗,然後與辣根過氧化物酶偶聯的驢抗山羊 IgG(1:1,000)孵育。 病毒分離 Vero E6 細胞單層保存在補充有 10% FCS 的 DMEM 中。 PCR 陽性樣品(在 200μl 緩衝液中)以 3,000–12,000g 梯度離心,上清液在 DMEM 中按 1:10 稀釋,然後加入 Vero E6 細胞。 在 37°C 孵育 1h 後,取出接種物並用含有 2% FCS 的新鮮 DMEM 代替。 將細胞在 37°C 下孵育 3 天,並每天檢查細胞病變效應。 雙劑量叄聯抗生素青黴素/鏈黴素/兩性黴素 (Gibco) 包含在所有組織培養基中(青黴素 200 IU ml−1,鏈黴素 0.2 mg ml−1,兩性黴素 0.5 μg ml−1)。 每個樣品進行叄個盲傳代。 每次傳代後,使用靶向 RdRP 或 S 基因的引物,通過 RT-PCR 檢查培養物上清液和細胞沉澱中是否存在病毒。 收集上清液 (10ml) 中的病毒粒子並使用 0.1% 甲醛固定 4h,然後使用 Ty90 轉子 (Beckman) 通過 20% 蔗糖墊 (5ml) 以 80,000g 超速離心 90min 進行濃縮。 將沉澱的病毒顆粒懸浮在 100 μl PBS 中,用 2% 磷鎢酸 (pH 7.0) 染色,並使用 Tecnai 透射電子顯微鏡 (FEI) 在 200 kV 下進行檢查。 免疫熒光法檢測病毒傳染性 擴展數據表 5 總結了用於本研究的細胞系及其培養條件。通過細胞病變效應 (CPE) 計數確定 Vero E6 細胞中的病毒滴度。 來自不同來源的細胞系和表達來自人類、果子狸或中國馬蹄蝠的 ACE2 的 HeLa 細胞在 24 孔板(康寧)的蓋玻片上生長,與蝙蝠 SL-CoV-WIV1 一起孵育,感染複數 = 10,持續 1 小時。 取出接種物並用 PBS 洗滌兩次並補充培養基。 無 ACE2 表達的 HeLa 細胞和 Vero E6 細胞分別用作陰性和陽性對照。 感染後 24 小時,用 PBS 洗滌細胞,並在 4°C 下用含 4% 甲醛的 PBS (pH 7.4) 固定 20 分鐘。 使用山羊抗人 ACE2 免疫球蛋白 (R&D Systems) 隨後使用 FITC 標記的驢抗山羊免疫球蛋白 (PTGLab) 檢測 ACE2 表達。 使用針對 SL-CoV Rp3 核衣殼蛋白的兔抗體,然後使用 Cy3 綴合的小鼠抗兔 IgG 檢測病毒複製。 細胞核用 DAPI 染色。 使用 FV1200 共聚焦顯微鏡 (Olympus) 檢查染色模式。 通過實時 RT-PCR 檢測病毒感染性 表達或不表達不同來源的 ACE2 的 Vero E6、A549、PK15、RSKT 和 HeLa 細胞接種 0.1 TCID50 WIV-1,並在 37°C 下孵育 1h。 除去接種物後,將細胞用含有1%FBS的培養基培養。 在 0、12、24 和 48h 收集上清液。 按照製造商的說明,使用病毒 RNA 迷你試劑盒(Qiagen)從 140μl 每種上清液中提取 RNA,並在 60μl 緩衝液 AVE(Qiagen)中洗脫。 在 ABI StepOne 系統上使用 TaqMan AgPath-ID 一步式 RT-PCR 試劑盒(Applied Biosystems)在 25μl 反應溷合物中對 RNA 進行定量,其中含有 4μl RNA,1 × RT-PCR 酶溷合物,1⟩×⟩RT-PCR 緩衝液、40pmol 正向引物 (5'-GTGGTGGTGACGGCAAAATG-3')、40pmol 反向引物 (5'-AAGTGAAGCTTCTGGGCCAG-3') 和 12pmol 探針 (5'-FAM-AAAGAGCTCAGCCCCAGATG-BHQ1-3')。 擴增參數為 50°C 10min、95°C 10min 以及 95°C 15s 和 60°C 20s 的 50 個循環。 來自純化的 WIV-1 原液的 RNA 稀釋液用作標準。 血清中和試驗 SARS 患者血清在 56°C 下滅活 30min,然後用於病毒中和測試。 血清從 1:10 開始稀釋,然後在 96 孔細胞板中連續兩倍稀釋至 1:40。 將每 100μl 血清稀釋液與 100μl 含有 100μl WIV1 TCID50 的病毒上清液溷合,並在 37℃ 下孵育 1h。 將溷合物一式叄份添加到鋪有單層 Vero E6 細胞的 96 孔細胞板的孔中,並在 37°C 下進一步孵育 2 天。 來自健康獻血者的血清在每個實驗中用作陰性對照。 接種後 2 天使用倒置顯微鏡觀察 CPE。 中和抗體滴度被讀取為完全抑制感染孔中 CPE 的血清的最高稀釋度。 中和試驗重複兩次。 重組分析 使用 Clustal X 將 SL-CoV Rs3367 或 RsSHC014 的全長基因組序列與選定的 SARS-CoV 和蝙蝠 SL-CoV 的基因組序列進行比對。使用重組檢測程序 (RDP) 4.0 初步掃描比對序列的重組事件(參考文獻 1)。 19). 由於 RDP 具有強 P 值 (<10–20),因此通過 Simplot 3.5.1 中實施的相似圖和引導掃描分析進一步研究了 RDP 提示的潛在重組事件。 Rs3367 或 RsSHC014 的主要和次要親本區域的系統發育起源是根據所選 SARS-CoV 和 SL-CoV 的主要和次要親本區域的基本 ORF 的串聯序列構建的。 使用 ClustalX 獨立比對了叄個估計斷點(20,827–26,553 和 26,554–28,685)之間的兩個基因組區域,並生成了 5,727 個鹼基對和 2,133 個鹼基對的兩個比對。 這兩個比對用於構建最大似然樹以更好地推斷片段父代。 本研究中的所有核苷酸編號均基於 Rs3367 基因組位置。 註冊碼 種質 基因庫/EMBL/DDBJ • KC880984 • KC881003 • KC881004 • KC881005 • KC881007 數據存款 叄種蝙蝠 SL-CoV 基因組、蝙蝠 SL-CoV RBD 和 R. sinicus ACE2 基因的序列已保存在 GenBank 中,登錄號為 KC881005–KC881007(分別來自 SL-CoV RsSHC014、Rs3367 和 W1V1 的基因組)、KC880984–KC881003( bat SL-CoV RBD 基因)和 KC881004(R. sinicus ACE2)。 |
|
|
 | |
|
 |
| 實用資訊 | |
|
|
| 一周點擊熱帖 | 更多>> |
| 一周回復熱帖 |
| 歷史上的今天:回復熱帖 |
| 2022: | 習近平聲稱專制體制(人壓迫人)將主宰 | |
| 2022: | 在一片掌聲中,加拿大的忑魯多宣布公民 | |
| 2021: | 大家看,六四就是這爛德行。毛賊當年喊 | |
| 2021: | 中國人民是中國共產黨政權的最大受害者 | |
| 2020: | 為何很多白人參加明尼阿波利斯黑人示威 | |
| 2020: | BREAKING:幼兒園老阿姨被茶館開了! | |
| 2019: | 評註《 六四改變了我們的人生軌跡-一些 | |
| 2019: | 我還是不當asshole吧。 | |
| 2018: | 一個公司是否倒閉,技術上直接取決於它 | |
| 2018: | 牛彈琴:中國人的好日子快到盡頭? | |