估算结论
1 CoVZC45通过任意12次变异演化出酶切位点的几率<1/10^52
2 CoVZC45一年内变异出酶切位点的概率<1/(10^36)
3 CoVZC45十年内变异出酶切位点的概率<1/(10^24)
4 CoVZC45一百年内变异出酶切位点的概率<1/(10^12)
5 CoVZC45一千年内变异出酶切位点的概率<25%
CoVZC45,指舟山蝙蝠类SARS冠状病毒bat-SL-CoVZC45(简称CoVZC45),它与SARS-CoV-2基因序列相似度87.5%,被公认为是与SARS-CoV-2亲缘关系最近,最有可能自然变异出SARS-CoV-2的冠状病毒。
2017年,南京军区军事医学研究所从采集于舟山的蝙蝠身上分离得到CoVZC45,2018年CoVZC45基因序列被上传至国际基因库。
引申结论
1. 由CoVZC45自然变异出酶切位点,进而演变成SARS-CoV-2是一个极度零概率事件。
如果,SARS-CoV-2是由与它亲缘关系最近的CoVZC45自然变异而来的,那么,没有酶切位点的CoVZC45,应在被发现的2017年至2019年11月之间,在2年时间内自然变异出SARS-CoV-2具有的酶切位点。由本文估算结果来看,这是一个零概率事件。
2. 由任何与SARS-CoV-2有足够亲缘关系(或相似度),且无酶切位点的病毒自然演变为SARS-CoV-2,是一个极度零概率事件。
此结论适用于SARS-CoV(SARS病毒),CoVZXC21(另一种舟山蝙蝠病毒),RaTG13(石正丽的所谓云南菊头蝠病毒,如果它确实存在的话)等β谱系冠状病毒。可参考文末“引申”部分。
准备
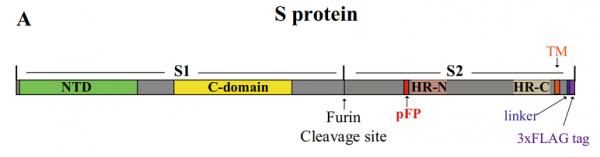
SARS-CoV-2的酶切位点,恰好位于刺突蛋白(Spike蛋白,或S蛋白)的两个亚基S1蛋白和S2蛋白的交界处,见上图。以下将此交界位置记为S1/S2。
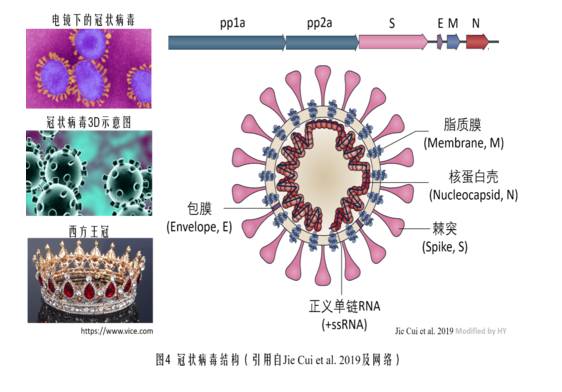
冠状病毒的构造图,病毒最外部的突起就是刺突蛋白(Spike,S),或棘突蛋白。
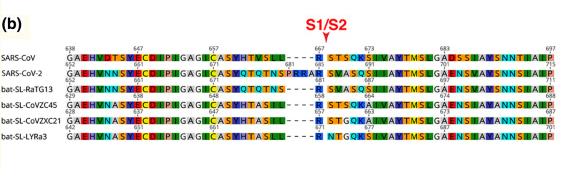
由上图可见,SARS-CoV-2在S1/S2位置新增了四个氨基酸(残基)“PRRA”,这四个氨基酸中的后三个“RRA”,与紧邻的“R”的组合,构成了SARS-CoV-2的furin酶切位点标志“RRAR”。图中第二行是SARS-CoV-2的氨基酸序列(或叫蛋白序列),第四行是CoVZC45的氨基酸序列。还可以参考下面这个图。

估算过程
A 估算CoVZC45通过任意12次变异演化出酶切位点的几率
舟山蝙蝠类SARS病毒CoVZC45一共有近3万个碱基对;
假设:
a) CoVZC45每变异一次,平均有N个位置会发生改变;
a) 每一个位置的变异方式有增、减、换3种,每种方式发生的机会均等;
则,在CoVZC45的任意一次变异中,S1/S2位置出现变异的概率是N/30000;
S1/S2位置出现的变异恰为新增一个碱基对的概率是(N/30000)*(1/3)=N/(30000*3)=N/(9*10^4);这里10^4表示10的4次方。
(简单起见,忽略在一次变异中,S1/S2位置同时新增2个以上碱基的情况)
(补注:已完成偏差估计,该忽略造成的偏差不超出估算结果上限的2倍,或者说,可能的几率上限不超过本估算结果上限的3倍。)
一个氨基酸对应三个碱基对。
通过CoVZC45的任意三次变异,在S1/S2处新增出三个碱基对的概率为(N/(9*10^4))^3=N^3/(9^3*10^12)=(N/9)^3*(1/10^12)
SARS-CoV-2的S1/S2处新增了四个氨基酸“PRRA”,第一个是P-脯氨酸,脯氨酸共有四种碱基对组合(每种组合含三个碱基对)。
RNA碱基对有U-A、C-G、A-U、G-C四种,三个碱基对的组合共有4*4*4=64种。在S1/S2处新增出的三个碱基对属于脯氨酸“P”的概率是4/64=1/16<1/10。
从而,通过任意三次变异,在S1/S2处新增出三个碱基对,且此三个碱基对属于脯氨酸“P”的概率<(N/9)^3*(1/10^12)* (1/10)=(N/9)^3*(1/10^13)
通过任意12次变异,在S1/S2处新增出全部四个氨基酸“PRRA”的概率
P<{(N/9)^3*(1/10^13)}^4=(N/9)^12*(1/10^52)
[补注:
可以优化模型与算法,涵盖一次变异产生多个碱基对的情况。新模型+算法的估算结果为:通过不超过12次变异,在S1/S2处新增出全部四个氨基酸“PRRA”的概率 P<3*(N/9)^12*(1/10^52)。
它是原估算结果的3倍级别,二者数量级一致。或者可以说,原估算的偏差不超出一个数量级。
]
如果N=1,可计算得P<(1/2.8)*(1/10^63),即小于10的负63次方;
如果N=9,则P<(1/10^52),即小于10的负52次方。
以上是一个基础性的估算。
下面进一步按不同时限估算。估算时,均取N=9;同时,假定CoVZC45一年内平均发生M=100次变异(参数M,N可能要根据相关研究进行调整)。
列出一个将反复用到的数值:12!=12*11*10*9。。。*1=479001600~=4.8*10^8>4*10^8>10^8。
B CoVZC45一年内变异出酶切位点的概率<1/(10^36)
CoVZC45一年内发生约100次变异;
从100次变异中取12次变异的组合数<100^12/12!
CoVZC45一年内通过某12次变异进化出酶切位点的概率
P1<(100^12/12!)*P<(10^24/10^8)/10^52=1/(10^36)
C CoVZC45十年内变异出酶切位点的概率<1/(10^24)
CoVZC45十年内约发生100*10=1000次变异。
从1000次变异中取12次变异的组合数<1000^12/12!
CoVZC45十年内进化出酶切位点的概率
P10<(1000^12/12!)/10^52<(10^36/10^8)/10^52=1/(10^24)
D CoVZC45 100年内变异出酶切位点的概率<1/(10^12)
CoVZC45 100年内约发生100*100=10000次变异。
从10000次变异中取12次变异的组合数<10000^12/12!
CoVZC45 100年内进化出酶切位点的概率
P100<(10000^12/12!)/10^52<(10^48/10^8)/10^52=1/(10^12)
E CoVZC45一千年内变异出酶切位点的概率<25%
CoVZC45 1000年内约发生100*1000=100000次变异。
从100000次变异中取12次变异的组合数<100000^12/12!
CoVZC45 1000年内进化出酶切位点的概率
P1000<(100000^12/12!)/10^52<(10^60/(4*10^8))/10^52=25%
引申
以上算法只依赖如下特征:
1. 估算对象是一个RNA单链冠状病毒;
2. 其S蛋白S1/S2位置无酶切位点氨基酸;
3. 基因序列长度约为3万个碱基对;
4. 变异频率约为100次/年,每次变异大约发生9个碱基对改变
所以,本文的估算及结论对以下病毒皆适用:
SARS-CoV,与SARS-CoV-2相似度78.7;
CoVZXC21(bat-SL-CoVZXC21),2015年采集于舟山,与SARS-CoV-2相似度87.3%;
RaTG13(Bat-CoV-RaTG13),石正丽2020年1月23日公布此病毒,称早在2013年即从云南菊头蝠粪便中分析得出,只有基因序列,没有病毒毒株,与SARS-CoV-2相似度约96.2%。
本文的估算和结论还可推广至所有与SARS-CoV-2有足够亲缘关系(或相似度),且无酶切位点的病毒(在SARS-CoV-2所在的β谱系,唯一具有酶切位点的是鼠肝炎冠状病毒)。









