补注:已完成本文的改进版本:“新冠RBD5个关键氨基酸抄袭、修改自rs3367”。改进版本对本文作了结论明确化、叙述简明化,它与本文并不冲突,但其结论价值及细节描述的准确性、严谨性皆高于本文。推荐您优先阅读改进版本。
本文完整标题:新冠刺突蛋白受体结合域决定感染人类能力的五个关键氨基酸是实验室定向选择的结果。
写作本文的目的是指出如下发现、事实或判断:
1。rs3367、rsSHC014、CoVZC45、Sars-Cov是四个与新冠(SARS-CoV-2)来源相关的重要冠状病毒。
2。新冠刺突蛋白受体结合域决定感染人类能力的五个关键氨基酸(残基)与rs3367、rsSHC014、Sars-Cov的对应关键氨基酸(残基)存在清晰的参照关系。
3。新冠五个关键氨基酸中,
第五个与Sars-Cov、rs3367及CoVZC45对应位置处的氨基酸相同,为酪氨酸Y;
第四个与rs3367对应位置处的氨基酸相同,为天门冬酰胺N;
第二个亦与rs3367对应位置处的氨基酸相同,为苯丙胺酸F;
第三个关键氨基酸是麸酰胺酸Q,它虽然与另外四个病毒对应位置处氨基酸都不相同,但rsSHC014的第三个关键氨基酸是精胺酸R,麸酰胺酸Q与精胺酸R是经常发生相向变异、相互替代的相关氨基酸;
第一个关键氨基酸是亮氨酸L(白氨酸),它也与另外四个病毒对应位置处氨基酸都不相同,但它与这些对应位置处氨基酸的电场、电离性质接近,五个病毒的第一个关键氨基酸要么是电荷分布均匀的非极性氨基酸,要么是不带电荷的极性氨基酸。
4。新冠的五个关键氨基酸中,有三个与rs3367的对应氨基酸相同,即新冠与rs3367的第五、第四、第二关键氨基酸相同。这样的重复度,无法用自然、偶然二字来解释。新冠的五个关键氨基酸,可能是基于rs3367的五个关键氨基酸,替换掉第三、第一个关键氨基酸之后得到的。
5。在五个关键位置上,新冠与Sars-Cov的对应氨基酸只在第五个位置上相同,在其它四个位置上两者的氨基酸都不相同,但援引相关论文,这四对相异氨基酸每一对的疏水性和静电极性都非常相似;
6。援引相关论文,新冠和Sars-Cov虽然有四个关键氨基酸的不同,但两者RBD(受体结合域)的3-D结构几乎没有明显差异,或者说,两者RBD的空间形态高度同构;而且,在与人类受体的接触界面上,两个RBD结构体的范德华力(一种分子间的电性引力)和静电作用效果非常相似;
7。基于第5点,新冠与SARS一样,也能与人类细胞受体ACE2发生强相互作用并良好结合,从而具有不亚于SARS的人体细胞感染能力。
8。新冠刺突蛋白RBD决定人类感染能力的五个关键氨基酸不是随机突变、无目的进化产生的,它们是基于明确的设计目标,参照、借鉴、复用特定病毒的关键氨基酸,人为确定的。用于新冠设计、改造的两个参照病毒rs3367、rsSHC014,都是罕见的具备跨物种传播能力,并可感染人体细胞的非常特殊的冠状病毒。
我要衷心感谢所有关注本文,参与讨论,指出文中错误、不当,予我批评、建议的读者朋友们。
同时,我也非常非常希望,科学、学术界的同仁、朋友们,能够注意到本文,对上述发现、事实、判断进行核查、确认、肯定或予以批驳、指错、纠正,以利于澄清真相,或在此基础上展开更深入的研究。
接下来,我们从一对姐妹病毒说起吧。
姐妹病毒:CoVZC45、CoVZXC21是已知的与新冠病毒(SARS-CoV-2)血缘关系最接近的两个冠状病毒。类SARS冠状病毒CoVZC45,2017年2月分离自舟山蝙蝠,基因序列2018年1月上传至国际基因库,全名为bat-SL-CoVZC45,GenBank
ID为:MG772933.1。它的姐姐类SARS冠状病毒CoVZXC21,2015年7月分离自舟山蝙蝠,与妹妹CoVZC45同一天上传至国际基因库,全名是bat-SL-CoVZXC21。
CoVZC45与新冠(SARS-CoV-2)基因组(基因序列)的相似度是87.5%(我用Blast比照的结果是89.12%),CoVZXC21与SARS-CoV-2的相似度是87.3%。这对姐妹彼此的相似度是97.48%。在绝大多数情况下,有关CoVZC45的内容同样适用于CoVZXC21。
相比之下,SARS-CoV(非典、萨斯)与SARS-CoV-2的相似度只有78.7%,比姐妹病毒低近10个百分点。
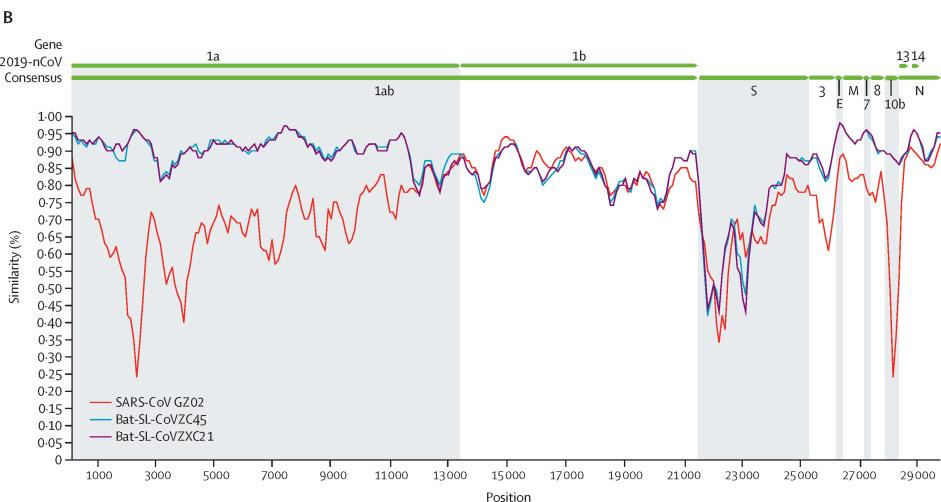
Sars-Cov(红)、CoVZC45(蓝)、CoVZXC21(紫)与Sars-Cov-2(2019-nCov)基因序列一致性对照图
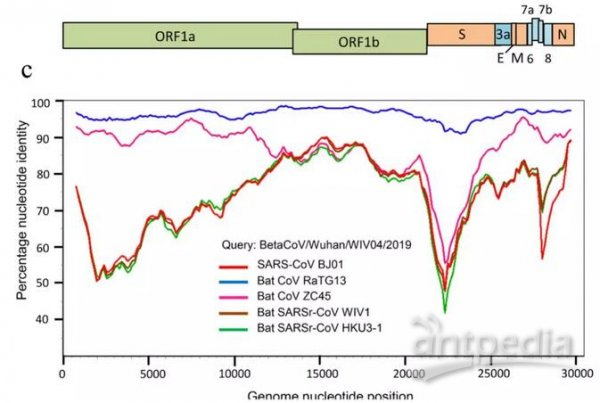
RaTG13、Sars-Cov、CoVZC45、WIV1等冠状病毒与Sars-Cov-2(2019-nCov)基因序列一致性对照图。2019-nCov是新冠的早期命名。
最近媒体频频提起的RaTG13不是新冠的近亲。RaTG13与SARS-CoV-2相似度虽然高达96.2%,但它只有基因序列,没有病毒毒株实证,它的基因序列是所谓“分析得出”的,很可能就是人为“创作”出来的,它没有严肃的证据价值和学术讨论价值;而且,就算它真实存在,它与新冠也没有时间进化关系,新冠不可能由它进化而来。关于RaTG13与新冠的进化无关性,南方医科大学2020年2月20日的论文“新型冠状病毒SARS-CoV-2的变异和进化分析”已经给出了明确说明。该论文的一个地址:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7086142/
如果新冠是自然进化产生的,它最可能是由CoVZC45或它的姐姐进化而来的,这也是新冠进化最近、最快捷的途径。但是这对姐妹病毒与新冠之间,仍然横着一道进化的天堑。观察前面两个插图,可以注意到,除RaTG13外,图中其它病毒与新冠在S蛋白位置都存在着鸿沟般的巨大(一致性)落差。姐妹病毒要进化为新冠,就必然完成诸多特定突变,消除它们与新冠在S蛋白上的显著差异。CoVZC45与新冠其它蛋白的相似度都不低于94%,但二者S1蛋白(S蛋白的前半部分)的相似度却猛降为69%。这决定了从CoVZC45到新冠的“演变”,是一个极不均衡、极为罕见、非常非常不自然的“自然进化”过程。而且,这一“自然进化”过程,居然只用了几年的时间就完成了(如果从姐妹病毒被发现的前后算起)。
自然界的冠状病毒约有一百种,绝大多数天然冠状病毒不能进入人体细胞,无法感染人类。大自然设置了冠状病毒的传播屏障:将S1蛋白决定宿主感染能力的重要部位设置为高度保守区域,这些部位的氨基酸通常不会发生突变。S1蛋白重要部位的稳定性使绝大多数冠状病毒在漫长的岁月里始终无法跨越S1蛋白的进化天堑,只能感染固定的动物宿主,难以跨物种传播,感染人类。
冠状病毒能否进入宿主细胞,感染宿主,取决于它有没有打开宿主细胞的“钥匙”。如果有匹配的细胞钥匙,它就能与宿主细胞表面的相关受体(相当于细胞锁)结合,继而进入宿主细胞。冠状病毒的细胞钥匙就是它的S蛋白,即Spike蛋白,也叫刺突蛋白。刺突蛋白包括相连的两个部分(两个亚基),最外部的是S1蛋白,S1蛋白与病毒包膜之间的是S2蛋白。S2蛋白也叫膜融合亚基(参与新冠病毒与细胞的膜融合),它相当于钥匙的手柄部分,S1蛋白是受体结合亚基,负责与细胞表面的受体结合,它相当于钥匙的齿面部分。S1蛋白中有一个区域叫做受体结合域(Receptor Binding Domain),简称RBD,是S1蛋白与细胞受体发生接触的部分,RBD就相当于齿面外侧的钥齿部位。RBD中有14个氨基酸与宿主细胞受体ACE2发生直接接触,这14个氨基酸中,有五个是至关重要的,它们决定刺突蛋白能否与ACE2结合,这五个关键氨基酸就是冠状病毒打开宿主细胞的关键钥齿。
注:ACE2,即Angiotensin Converting Enzyme2,血管紧张素转换酶2,是细胞表面的一个膜蛋白,广泛存在于脊椎动物的呼吸、消化、血液、泌尿、生殖、视觉等系统的器官、组织内。ACE2可介导与之发生结合的病毒进入它所附着的细胞内部。ACE2也是肾素-血管紧张素系统(RAS)中的一个关键酶;hACE2,即human ACE2,人体细胞ACE2受体。
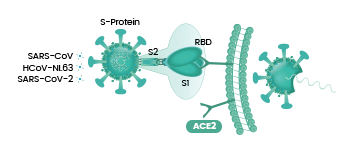
S1蛋白与宿主细胞受体(如ACE2)结合成功后,病毒可进而通过“胞吞”(如Sars-Cov)或“膜融合”(如Sars-Cov-2)两种方式之一进入宿主细胞。
可感染人类,并产生疾病症状的冠状病毒已知有7种,其中包括SARS(Sars-Cov)和新冠(Sars-Cov-2)。除这7种病毒外,还有两个很特别的冠状病毒:rsSHC014和rs3367,它们也能与hACE2(人体细胞ACE2)结合,进入人体细胞,但不会使人发病,且对人体无害。
RsSHC014,也称作SHC014-Cov或SHC014病毒,GenBank ID为: KC881005.1。
rs3367,也叫WIV1病毒(SL-CoV-WIV1),GenBank ID为: KC881006.1。前面第二个贴图中出现过这个病毒。
RsSHC014、rs3367都搜集自云南昆明中华菊头蝠(亦称中华马蹄蝠)的肠道与粪便样本,其病毒株分离获得时间同为2011年4月,它们的基因序列上传至国际基因库的时间同为2013年4月。
RsSHC014、rs3367与SARS-CoV的基因组相似度均为95%,被认为极可能是SARS-CoV的自然进化来源。rs3367的RBD(Receptor Binding Domain,受体结合域)与SARS-CoV接近程度相当高,在RBD与宿主受体ACE2直接接触的14个位点中,rs3367与SARS-CoV在11个位点上氨基酸相同,虽然另有三个位点氨基酸不同,但rs3367仍能与hACE2(Human ACE2)良好结合并进入人类细胞。相比之下,在RBD14个直接接触位点中,RsSHC014与SARS-CoV在7个位点上氨基酸不同,但2015年秋拉尔夫·S·巴里克、Vineet D Menachery、石正丽、葛行义等人的著名实验证明,虽然与Sars-Cov有一半RBD接触位点不同,但RsSHC014的刺突蛋白仍然能与hACE2良好结合。
rs3367是一种极不寻常的的冠状病毒,它具备强大的跨物种传播能力,它能感染的动物包括:人类、果子狸、貉、恒河猴、雪貂、水鼬、猫、蝙蝠、老鼠。
rs3367和RsSHC014虽然鲜为普通民众所知,但在冠状病毒研究领域,它们绝非无名之辈,在病毒跨物种传播研究领域,它们更是极为重要的研究对象。它们的重要性主要在于其S(Spike)蛋白,即刺突蛋白。对它们的研究在2016年春之前就已经相当深入,并已取得了多个重大成果。我列举几个与rs3367或RsSHC014紧密相关的研究工作。
2013年10月,
石正丽、Peter Daszak、葛行义等人发表了一篇重要论文:”Isolation and characterization of a
bat SARS-like coronavirus that uses the ACE2
receptor”,论文地址:https://www.nature.com/articles/nature12711
该论文对rs3367(实验使用的SL-CoV-WIV1与rs3367有99.9%的序列一致性,本文将它们视作同一病毒)进行了深入研究,证实了rs3367或WIV1具有跨物种传播能力:可与细胞的ACE2受体结合,并以此进入人类、果子狸、中华马蹄蝠的细胞。该论文证实了,自然界中存在与Sars-Cov一样可跨物种传播的类Sars冠状病毒,它被认为是Sars-Cov起源于自然界的有力证明。
2015年11月,Ralph S Baric、Vineet D Menachery、石正丽、葛行义等人发表了著名的病毒合成实验论文,参与论文的包括美国、中国、瑞士三国的大学、研究机构、政府部门。论文地址:
https://www.nature.com/articles/nm.3985
这次研究最重要的实验成果是,用RsSHC014的刺突蛋白和Sars-Cov-MA15(一种实验室多次传代培育的可感染并使小鼠致病的Sars-Cov病毒变异体)的病毒主干合成了一种人工改造病毒,把不能使人、鼠发病的RsSHC014改造成了可使人、鼠致病,可使实验小鼠致死的危险嵌合病毒SHC014-MA15。这次实验为改造致病性病毒开创了道路,SHC014-MA15相当于Sars-Cov-2的一个早期原型,它们的基本原理是相同的,区别在于,Sars-Cov-2比SHC014-MA15技术上更复杂,功能更丰富、强大,它还有一定的“自然进化”伪装性。
2016年3月,Ralph S. Baric、Vineet D. Menachery等人又发表了一篇研究rs3367(WIV1)的论文。论文地址:https://www.pnas.org/content/113/11/3048
这一次,研究者们用rs3367的刺突蛋白与Sars-Cov-MA15合成了另外一种嵌合病毒WIV1-MA15。实验证明,WIV1-MA15能够感染人体细胞,在呼吸道、体内大量复制并使人发病,但症状不及Sars严重。这是Ralph
S. Baric、Vineet D.
Menachery等人又一次使用嵌合方式改造出人类致病病毒,他们再一次研究了实验室改造病毒对人类的致病能力,以及对该病毒应用抗体治疗的效果。
上述两篇论文的第一作者都是Vineet D. Menachery,通讯作者都是Ralph S. Baric。二人都是北卡罗来纳大学教堂山分校的病毒学家、生物医药学家,同时,他们也都是瑞得西韦(Remdesivir)的研发人员。
从上述两篇病毒嵌合相关的论文我们可以得到一些启示。实验室改造病毒,使之跨物种传播的首要环节是换钥匙或配钥匙,也就是把不能打开人体细胞(即不能与人类细胞受体结合)的刺突蛋白(如CoVZC45或CoVZXC21的刺突蛋白),替换或改造成可打开人体细胞的刺突蛋白(如Sars-Cov、rs3367、rsSHC014的刺突蛋白)。要得到一把好用的“钥匙”,可以把钥匙整个换掉,也可以只换齿面或只修磨钥齿。可以只处理关键的几个钥齿,也就是改造刺突蛋白RBD(Receptor
Binding Domain)中的五个关键氨基酸。
作了很长的铺垫,现在可以说新冠的五个关键氨基酸与rs3367、rsSHC014、Sars-Cov、CoVZC45的关键氨基酸之间的关联了。
新冠刺突蛋白RBD的五个关键氨基酸依次为:
亮胺酸L(451),苯丙胺酸F(482),麸酰胺酸Q(489),天门冬酰胺N(497)和酪胺酸Y(501)。
氨基酸名称、缩写字母后的数字是该氨基酸在刺突蛋白的氨基酸序列中的序号。
注:上述关键氨基酸序号是基于(氨基酸序列)长度为1269的新冠刺突蛋白而言的,如果选用长度为1273的新冠刺突蛋白,那么五个关键氨基酸依次为:
亮胺酸L(455),苯丙胺酸F(486),麸酰胺酸Q(493),天门冬酰胺N(501),酪胺酸Y(505)。
Spike_1269与Spike_1273的关键氨基酸没有区别,只是氨基酸序号相差4。在Spike_1269的氨基酸141、142之间插入四个氨基酸LGVY,就得到了Spike_1273,二者其它对应位置的氨基酸完全相同,使用Spike_1269或Spike_1273,对本文来说是等价的。
CoVZC45对应位置上的五个关键氨基酸为:丝氨酸S(446),空缺,丝氨酸S(470),缬胺酸V(478),酪氨酸Y(482)。这二个病毒关键氨基酸的对照情况如下图:

注:CoVZC45的Spike蛋白NCBI地址
https://www.ncbi.nlm.nih.gov/protein/AVP78031.1
不难发现,CoVZC45与新冠只有第五个关键氨基酸相同,同为酪氨酸Y,而且,CoVZC45第二个关键钥齿氨基酸缺失!如果CoVZC45要进化为新冠,它必须:
在第二个关键位置处突变增生出一个苯丙胺酸F;
将第一个关键钥齿-丝氨酸S(446)突变、进化为亮胺酸L;
将第三个关键钥齿-丝氨酸S(470)突变、进化为麸酰胺酸Q;
将第四个关键钥齿-缬胺酸V(478)突变、进化为天门冬酰胺N;
保持第五个关键钥齿-酪胺酸Y不变。
另外,可以看到,两个病毒S1蛋白关键钥齿有一定的序号错位,这是CoVZC45的刺突蛋白氨基酸序列相对较短造成的,这需要CoVZC45变异增生出一定数量的氨基酸,消除氨基酸序列长度差异及氨基酸序号错位,这无疑将加大CoVZC45进化为新冠的难度。
下面看看Sars-Cov、rs3367、rsSHC014这三个病毒的关键钥齿情况:
Sars-Cov的五个关键氨基酸为:酪氨酸Y(442),亮胺酸L(472),天门冬酰胺N(479),苏胺酸T(487),酪氨酸Y(491);
rs3367的五个关键氨基酸为:丝氨酸S(443),苯丙胺酸F(473),天门冬酰胺N(480),天门冬酰胺N(488),酪胺酸Y(492)。
rsSHC014的五个关键氨基酸为:色胺酸W(443),脯胺酸P(473),精胺酸R(480),丙胺酸A(488), 组胺酸H(492)。
在上述氨基酸中,精胺酸R、麸酰胺酸Q是经常发生相向变异、相互替代的相关氨基酸;组胺酸H、酪胺酸Y也是经常发生相向变异、相互替代的相关氨基酸。
注:
Sars-Cov的Spike蛋白NCBI地址
https://www.ncbi.nlm.nih.gov/protein/AAR07630.1
rs3367的Spike蛋白NCBI地址
https://www.ncbi.nlm.nih.gov/protein/AGZ48818.1
rsSHC014的Spike蛋白NCBI地址
https://www.ncbi.nlm.nih.gov/protein/AGZ48806.1
将五个病毒的关键钥齿氨基酸情况汇总于下表。
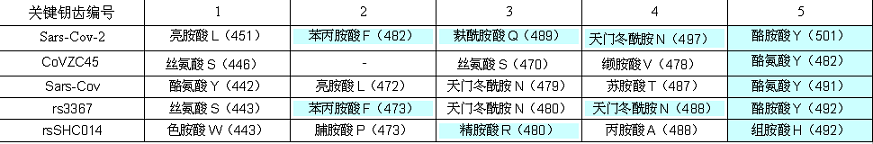
从上表容易观察到:
1。在第五关键钥齿位置,前四个病毒的氨基酸都是酪胺酸Y,rsSHC014的相应氨基酸虽然是组胺酸H,但它与酪胺酸Y是经常发生相互变异、替代的相关氨基酸。这也许表明,在五个关键钥齿中,第五钥齿最核心,也最保守,是决定人类感染能力的最重要的一个钥齿氨基酸;
2。新冠的第五关键钥齿、第四关键钥齿、第二关键钥齿都与rs3367的对应关键钥齿相同,也就是说,新冠有三个关键钥齿和rs3367相同;
3。新冠的第三关键钥齿麸酰胺酸Q虽然与其它四个病毒都不相同,但它与rsSHC014的第三关键钥齿精胺酸R是经常发生相互替代的氨基酸。
新冠的第一关键钥齿亮胺酸L的来历渊源还没有完全调查清楚。第一关键钥齿可能没有特别的参照来源,它可能是基于另外四个关键钥齿,从某一候选范围中选择、试验出来的。如果以某一候选氨基酸+其它四个关键钥齿的组合构造的RBD与人体细胞受体适配,那么该候选氨基酸就可以作为第一关键钥齿。
粗略考虑一下第一关键钥齿的候选范围。
五个病毒中,只有CoVZC45的刺突蛋白打不开人体细胞,其它四种病毒的刺突蛋白对人体细胞(受体锁)都有效。从上图第二列可见,除CoVZC45外,其它四个病毒的第一关键钥齿,相应的四个氨基酸依次是:亮胺酸L(Sars-Cov-2)、酪胺酸Y(Sars-Cov)、丝氨酸S(rs3367)、色胺酸W(rsSHC014)。
这四种氨基酸中,亮氨酸L,色氨酸W是电荷分布均匀的非极性氨基酸,丝氨酸S,酪氨酸Y则是不带电荷的极性氨基酸。也就是说,这四种能感染人的病毒,它们的关键钥齿氨基酸都是电荷分布均匀或电荷中性的,它们的电场、电离属性接近。
因此,可以先从电荷分布均匀或不带电荷的氨基酸中选择候选的新冠第一关键钥齿,符合这一条件的氨基酸共15种。这个范围可能偏大,可以考虑其它条件,先在更小的范围内选择。如,以Sars-Cov的第一关键钥齿-酪胺酸Y为基准,挑选与它的范德华半径差<=20,且疏水性、酸碱性相同的氨基酸。符合这一条件的氨基酸有4种:甲硫胺酸、亮胺酸、异亮胺酸、苯丙胺酸,最终入选新冠第一关键钥齿的亮胺酸也在其中。通过上述思路来挑选新冠第一关键钥齿,从而确定新冠的全部五个关键钥齿是完全可行的。
如果新冠是由CoVZC45自然进化产生的,那么,CoVZC45在五个关键钥齿部位的进化情况是:
在第二个关键钥齿处突变增生出与rs3367对应位置相同的苯丙胺酸F;
第四关键钥齿缬胺酸V(478)突变、进化为与rs3367第四关键钥齿相同的天门冬酰胺N;
第三关键钥齿丝氨酸S(470)突变、进化为与rsSHC014第三关键钥齿精胺酸R可互相替换的麸酰胺酸Q;
第一关键钥齿丝氨酸S(446)突变、进化为亮胺酸L;
保持第五关键钥齿酪胺酸Y(482)不变(但它的位置序号将变为501,它的前面将有若干新增生的氨基酸)。
上述四项突变、进化都发生在刺突蛋白的高度保守区域,通常,高度保守区域内的氨基酸是很少发生突变的。不论其它位置的突变,仅四个关键钥齿的突变,及其位置序号的准确改变,就是几十年、几百年、几千年。。。所无法完成的,更何况这四个关键钥齿不是随机突变成什么都行,而是各自都要变化成确定的氨基酸。
还有,舟山群岛冠状病毒CoVZC45,怎么偏巧就进化出了和云南冠状病毒rs3367、rsSHC014相同或可替换的关键钥齿?而这两个病毒,恰巧又是病毒学家们反复研究、实验过,对其了如指掌的病毒。该如何解释这些巧合?这样偶然又偶然的事,自然发生的几率有多大?
新冠决定人类感染能力的五个关键氨基酸不是随机突变、进化产生的,它们是基于明确的参照物rs3367、rsSHC014,人为设计,定向挑选、试验的结果。

简单概括一下,新冠五个关键钥齿可能的确定过程:
1。以参照病毒rs3367的五个关键钥齿为新冠关键钥齿的初始原型,并保持第五、第四、第二关键钥齿不变;
2。尝试替换掉第三关键钥齿-天门冬酰胺N。实验发现,参照病毒rsSHC014的第三关键钥齿精胺酸R,以及精胺酸R的常发变异替代者麸酰胺酸Q均可作为替代者,换入新的第三关键钥齿后,RBD仍能与人体细胞受体良好结合。以麸酰胺酸Q为新冠的第三关键钥齿。
3。尝试替换掉第一关键钥齿-丝氨酸S。如前所述,在适当的范围内选择、试验候选氨基酸。实验发现,以亮胺酸L替换丝氨酸S后,RBD仍能与人体细胞受体良好结合。以亮胺酸L为新冠的第一关键钥齿。
至此,新冠的五个关键钥齿都已确定。
以上只是对新冠5关键钥齿确定过程的粗略推测,它可能与实际的确定过程存在某些出入,比如,第三关键钥齿、第一关键钥齿可能并非一先一后逐个确定的,它们可能是从若干双候选组合中同时选出、确定的。
对比Sars-Cov 、新冠的五个关键氨基酸、RBD及hACE2结合效果:
1。新冠与Sars-Cov的五个关键钥齿氨基酸只有第五个相同,其它四个都不相同,但这四对相异氨基酸每一对的疏水性和静电极性都非常相似;
2。虽然有四个关键氨基酸不同,但两者RBD(受体结合域)的3-D结构几乎没有明显差异,或者说,两者RBD的空间形态高度同构;在与人类受体的接触界面上,两个RBD结构体的范德华力(一种分子间的电性引力)和静电作用效果非常相似;
3。与SARS一样,新冠的RBD也能与人类细胞受体ACE2发生强相互作用并良好结合,新冠具有不亚于SARS的人体细胞感染能力。
SARS-Cov与Sars-Cov-2刺突蛋白关键氨基酸异同,及空间结构示意图
与新冠、Sars-Cov二病毒RBD空间结构及受体结合能力有关的两篇论文。
论文一。2020年1月21日,来自中国科学院上海巴斯德研究所等单位的徐心恬、陈萍、王靖方等学者在SCIENCE
CHINA Life
Sciences(《中国科学:生命科学》)上发表了一篇论文,题为“源于武汉爆发的新型冠状病毒的进化及其棘突蛋白对人类传播风险的建模”(Evolution
of the novel coronavirus from the ongoing Wuhan outbreak and modeling
of its spike protein for risk of human transmission)。论文地址为:
https://link.springer.com/content/pdf/10.1007/s11427-020-1637-5.pdf
论文指出:
a)尽管新冠与SARS-CoV二者S蛋白的总体同源性较低,但二者的RBD区域却有较高的同源性;
b)令我们惊讶的是,尽管替换了五个重要的界面氨基酸残基中的四个,但新冠仍与人体ACE2有着显著的结合亲和力;
c)进一步观察发现,新冠的S蛋白和SARS-CoV的S蛋白在RBD域具有几乎相同的三维结构,在相互作用界面上保持了相似的范德华力和静电性质。
论文二。2020年2月19日,美国德克萨斯大学奥斯汀分校Jason
S McLellan团队,及国家过敏和传染病研究所的研究人员在Science杂志上联合发表了题为“Cryo-EM Structure of
the 2019-nCoV Spike in the Prefusion Conformation”的论文。论文地址为:
https://science.sciencemag.org/content/367/6483/1260
论文指出:
a) 2019-nCoV与SARS-CoV的RBD存在一定的构象差异,但当按某一角度对齐时,二者就呈现出较高程度的结构同源性。
b) 两种病毒的S1蛋白与ACE2的复合体非常相似;
c) 2019-nCoV的S1蛋白与ACE2的结合亲和力是Sars-Cov的10-20倍。
最后,我们将关键钥齿的确定过程延展,设想一下新冠实验室改造的可能流程:
将CoVZC45的主干与Sar-Cov(或rs3367、rsSHC014)的(扩展)RBD嵌合,得到新冠的原型病毒。原型病毒已具备感染人的能力;
参照rs3367、rsSHC014的关键钥齿氨基酸,修改原型病毒的关键钥齿氨基酸,确定新冠最终的五个关键钥齿氨基酸;
参照rs3367、rsSHC014、Sars-Cov等病毒的RBD及S1蛋白,编辑新冠RBD及S1蛋白的其它位置,模糊嵌合操作痕迹;
参照Sar-Cov、HIV-1、埃博拉、HEV71等具备特殊功能的病毒,进行其它功能增益(Gain-of-Function)改造,包括复制能力、扩散能力增强,设计、实现免疫破坏力、抗免疫能力、免疫干扰能力等等;
进行其它基因编辑及实验室痕迹模糊化处理。
关于新冠的功能增益,我去年的一篇文章“新冠君,你额头的Lab金印洗得掉么?”的第3~第6部分中有一些总结,有兴趣的读者可参考。
(正文完)













